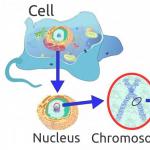
डीएनए अणु में दो स्ट्रैंड होते हैं जो एक डबल हेलिक्स बनाते हैं। इसकी संरचना को सबसे पहले 1953 में फ्रांसिस क्रिक और जेम्स वॉटसन ने समझा था।
सबसे पहले, डीएनए अणु, जिसमें एक दूसरे के चारों ओर मुड़ी हुई न्यूक्लियोटाइड श्रृंखलाओं की एक जोड़ी शामिल थी, ने इस सवाल को जन्म दिया कि इसका यह विशेष आकार क्यों है। वैज्ञानिक इस घटना को संपूरकता कहते हैं, जिसका अर्थ है कि केवल कुछ न्यूक्लियोटाइड ही इसके स्ट्रैंड में एक दूसरे के विपरीत पाए जा सकते हैं। उदाहरण के लिए, एडेनिन हमेशा थाइमिन के विपरीत होता है, और गुआनिन हमेशा साइटोसिन के विपरीत होता है। डीएनए अणु के इन न्यूक्लियोटाइड्स को पूरक कहा जाता है।
योजनाबद्ध रूप से इसे इस प्रकार दर्शाया गया है:
टी-ए
सी - जी
ये जोड़े एक रासायनिक न्यूक्लियोटाइड बंधन बनाते हैं, जो अमीनो एसिड का क्रम निर्धारित करता है। पहले मामले में यह थोड़ा कमजोर है. C और G के बीच संबंध अधिक मजबूत है. गैर-पूरक न्यूक्लियोटाइड एक दूसरे के साथ जोड़े नहीं बनाते हैं।
भवन के बारे में
अतः डीएनए अणु की संरचना विशेष होती है। इसका यह आकार एक कारण से है: तथ्य यह है कि न्यूक्लियोटाइड की संख्या बहुत बड़ी है, और लंबी श्रृंखलाओं को समायोजित करने के लिए बहुत अधिक जगह की आवश्यकता होती है। यही कारण है कि जंजीरों की विशेषता एक सर्पिल मोड़ है। इस घटना को स्पाइरलाइजेशन कहा जाता है, यह धागों को लगभग पांच से छह गुना छोटा करने की अनुमति देता है।
शरीर इस प्रकार के कुछ अणुओं का बहुत सक्रिय रूप से उपयोग करता है, अन्य का शायद ही कभी। उत्तरार्द्ध, स्पाइरलाइज़ेशन के अलावा, सुपरस्पिरलाइज़ेशन जैसी "कॉम्पैक्ट पैकेजिंग" से भी गुजरता है। और फिर डीएनए अणु की लंबाई 25-30 गुना कम हो जाती है।

किसी अणु की "पैकेजिंग" क्या है?
सुपरकोइलिंग की प्रक्रिया में हिस्टोन प्रोटीन शामिल होते हैं। उनकी संरचना और स्वरूप धागे के स्पूल या छड़ी की तरह है। उन पर सर्पिल धागे लपेटे जाते हैं, जो तुरंत "कॉम्पैक्टली पैक" हो जाते हैं और बहुत कम जगह लेते हैं। जब एक या दूसरे धागे का उपयोग करने की आवश्यकता होती है, तो इसे स्पूल से खोल दिया जाता है, उदाहरण के लिए, हिस्टोन प्रोटीन, और हेलिक्स दो समानांतर श्रृंखलाओं में खुल जाता है। जब डीएनए अणु इस अवस्था में होता है, तो उससे आवश्यक आनुवंशिक डेटा पढ़ा जा सकता है। हालांकि, एक शर्त है। जानकारी प्राप्त करना तभी संभव है जब डीएनए अणु की संरचना बिना मुड़े हुए आकार की हो। पढ़ने के लिए सुलभ क्रोमोसोम को यूक्रोमैटिन कहा जाता है, और यदि वे सुपरकोइल्ड हैं, तो वे पहले से ही हेटरोक्रोमैटिन हैं।
न्यूक्लिक एसिड
न्यूक्लिक एसिड, प्रोटीन की तरह, बायोपॉलिमर हैं। मुख्य कार्य वंशानुगत (आनुवंशिक जानकारी) का भंडारण, कार्यान्वयन और प्रसारण है। वे दो प्रकार में आते हैं: डीएनए और आरएनए (डीऑक्सीराइबोन्यूक्लिक और राइबोन्यूक्लिक)। उनमें मोनोमर्स न्यूक्लियोटाइड होते हैं, जिनमें से प्रत्येक में एक फॉस्फोरिक एसिड अवशेष, एक पांच-कार्बन चीनी (डीऑक्सीराइबोज/राइबोज) और एक नाइट्रोजनस बेस होता है। डीएनए कोड में 4 प्रकार के न्यूक्लियोटाइड शामिल हैं - एडेनिन (ए) / गुआनिन (जी) / साइटोसिन (सी) / थाइमिन (टी)। उनमें मौजूद नाइट्रोजन आधार में भिन्नता होती है।
एक डीएनए अणु में, न्यूक्लियोटाइड की संख्या बहुत बड़ी हो सकती है - कई हजार से लेकर दसियों और लाखों तक। ऐसे विशाल अणुओं की जांच इलेक्ट्रॉन माइक्रोस्कोप से की जा सकती है। इस मामले में, आप पॉलीन्यूक्लियोटाइड स्ट्रैंड्स की एक दोहरी श्रृंखला देख पाएंगे, जो न्यूक्लियोटाइड्स के नाइट्रोजनस आधारों के हाइड्रोजन बांड द्वारा एक दूसरे से जुड़े हुए हैं।

अनुसंधान
शोध के दौरान वैज्ञानिकों ने पाया कि विभिन्न जीवित जीवों में डीएनए अणुओं के प्रकार भिन्न-भिन्न होते हैं। यह भी पाया गया कि एक श्रृंखला का गुआनिन केवल साइटोसिन से बंध सकता है, और थाइमिन एडेनिन से। एक श्रृंखला में न्यूक्लियोटाइड की व्यवस्था सख्ती से समानांतर से मेल खाती है। पॉलीन्यूक्लियोटाइड्स की इस पूरकता के लिए धन्यवाद, डीएनए अणु दोगुना और आत्म-प्रजनन करने में सक्षम है। लेकिन सबसे पहले, पूरक श्रृंखलाएं, विशेष एंजाइमों के प्रभाव में जो युग्मित न्यूक्लियोटाइड को नष्ट करती हैं, अलग हो जाती हैं, और फिर उनमें से प्रत्येक में लापता श्रृंखला का संश्लेषण शुरू होता है। ऐसा प्रत्येक कोशिका में बड़ी मात्रा में मौजूद मुक्त न्यूक्लियोटाइड के कारण होता है। इसके परिणामस्वरूप, "माँ अणु" के बजाय, दो "बेटी" अणु बनते हैं, जो संरचना और संरचना में समान होते हैं, और डीएनए कोड मूल बन जाता है। यह प्रक्रिया कोशिका विभाजन का अग्रदूत है। यह मातृ कोशिकाओं से बेटी कोशिकाओं, साथ ही बाद की सभी पीढ़ियों तक सभी वंशानुगत डेटा के संचरण को सुनिश्चित करता है।
जीन कोड कैसे पढ़ा जाता है?
आज, न केवल डीएनए अणु के द्रव्यमान की गणना की जाती है - बल्कि अधिक जटिल डेटा का पता लगाना भी संभव है जो पहले वैज्ञानिकों के लिए दुर्गम था। उदाहरण के लिए, आप इस बारे में जानकारी पढ़ सकते हैं कि कोई जीव अपनी कोशिका का उपयोग कैसे करता है। बेशक, सबसे पहले यह जानकारी एन्कोडेड रूप में होती है और इसमें एक निश्चित मैट्रिक्स का रूप होता है, और इसलिए इसे एक विशेष वाहक तक पहुंचाया जाना चाहिए, जो कि आरएनए है। राइबोन्यूक्लिक एसिड परमाणु झिल्ली के माध्यम से कोशिका में प्रवेश करने और अंदर एन्कोडेड जानकारी को पढ़ने में सक्षम है। इस प्रकार, आरएनए नाभिक से कोशिका तक छिपे हुए डेटा का वाहक है, और यह डीएनए से इस मायने में भिन्न है कि इसमें डीऑक्सीराइबोज़ के बजाय राइबोज़ और थाइमिन के बजाय यूरैसिल होता है। इसके अलावा, आरएनए एकल-फंसे हुए है।

आरएनए संश्लेषण
डीएनए के गहन विश्लेषण से पता चला है कि आरएनए नाभिक छोड़ने के बाद, साइटोप्लाज्म में प्रवेश करता है, जहां इसे राइबोसोम (विशेष एंजाइम सिस्टम) में मैट्रिक्स के रूप में एकीकृत किया जा सकता है। प्राप्त जानकारी से निर्देशित होकर, वे प्रोटीन अमीनो एसिड के उचित अनुक्रम को संश्लेषित कर सकते हैं। राइबोसोम त्रिक कोड से सीखता है कि किस प्रकार के कार्बनिक यौगिक को बनाने वाली प्रोटीन श्रृंखला से जोड़ा जाना चाहिए। प्रत्येक अमीनो एसिड का अपना विशिष्ट त्रिक होता है, जो इसे एनकोड करता है।
श्रृंखला का निर्माण पूरा होने के बाद, यह एक विशिष्ट स्थानिक रूप प्राप्त कर लेता है और एक प्रोटीन में बदल जाता है जो अपने हार्मोनल, निर्माण, एंजाइमेटिक और अन्य कार्य करने में सक्षम होता है। किसी भी जीव के लिए यह एक जीन उत्पाद है। इसी से जीन के सभी प्रकार के गुण, गुण और अभिव्यक्तियाँ निर्धारित होती हैं।

जीन
अनुक्रमण प्रक्रियाएं मुख्य रूप से यह जानकारी प्राप्त करने के लिए विकसित की गईं कि डीएनए अणु की संरचना में कितने जीन हैं। और, हालाँकि शोध ने वैज्ञानिकों को इस मामले में बड़ी प्रगति करने की अनुमति दी है, लेकिन उनकी सटीक संख्या जानना अभी भी संभव नहीं है।
कुछ साल पहले यह माना गया था कि डीएनए अणुओं में लगभग 100 हजार जीन होते हैं। कुछ समय बाद यह आंकड़ा घटकर 80 हजार हो गया और 1998 में आनुवंशिकीविदों ने बताया कि एक डीएनए में केवल 50 हजार जीन मौजूद होते हैं, जो कुल डीएनए लंबाई का केवल 3% है। लेकिन आनुवंशिकीविदों के नवीनतम निष्कर्ष चौंकाने वाले थे। अब उनका दावा है कि जीनोम में इनमें से 25-40 हजार इकाइयां शामिल हैं। यह पता चला है कि क्रोमोसोमल डीएनए का केवल 1.5% प्रोटीन कोडिंग के लिए जिम्मेदार है।
शोध यहीं नहीं रुका। जेनेटिक इंजीनियरिंग विशेषज्ञों की एक समानांतर टीम ने पाया कि एक अणु में जीन की संख्या ठीक 32 हजार है। जैसा कि आप देख सकते हैं, निश्चित उत्तर पाना अभी भी असंभव है। बहुत सारे विरोधाभास हैं. सभी शोधकर्ता केवल अपने परिणामों पर भरोसा करते हैं।
क्या वहां विकास हुआ था?
इस तथ्य के बावजूद कि अणु के विकास का कोई सबूत नहीं है (चूंकि डीएनए अणु की संरचना नाजुक और आकार में छोटी है), वैज्ञानिकों ने अभी भी एक धारणा बनाई है। प्रयोगशाला के आंकड़ों के आधार पर, उन्होंने निम्नलिखित संस्करण को आवाज दी: अपनी उपस्थिति के प्रारंभिक चरण में, अणु में एक सरल स्व-प्रतिकृति पेप्टाइड का रूप था, जिसमें प्राचीन महासागरों में पाए जाने वाले 32 अमीनो एसिड शामिल थे।
स्व-प्रतिकृति के बाद, प्राकृतिक चयन की शक्तियों के कारण, अणुओं ने बाहरी तत्वों से खुद को बचाने की क्षमता हासिल कर ली। वे अधिक समय तक जीवित रहने लगे और बड़ी मात्रा में प्रजनन करने लगे। जिन अणुओं ने खुद को लिपिड बुलबुले में पाया, उनके पास खुद को पुन: उत्पन्न करने का हर मौका था। क्रमिक चक्रों की एक श्रृंखला के परिणामस्वरूप, लिपिड बुलबुले ने कोशिका झिल्ली का रूप प्राप्त कर लिया, और फिर - प्रसिद्ध कणों का। यह ध्यान दिया जाना चाहिए कि आज डीएनए अणु का कोई भी भाग एक जटिल और स्पष्ट रूप से कार्य करने वाली संरचना है, जिसकी सभी विशेषताओं का वैज्ञानिकों ने अभी तक पूरी तरह से अध्ययन नहीं किया है।

आधुनिक दुनिया
हाल ही में इजराइल के वैज्ञानिकों ने एक ऐसा कंप्यूटर विकसित किया है जो प्रति सेकंड खरबों ऑपरेशन कर सकता है। आज यह पृथ्वी पर सबसे तेज़ कार है। पूरा रहस्य यह है कि नवोन्मेषी उपकरण डीएनए द्वारा संचालित है। प्रोफेसरों का कहना है कि निकट भविष्य में ऐसे कंप्यूटर ऊर्जा भी पैदा करने में सक्षम होंगे।
एक साल पहले, रेहोवोट (इज़राइल) में वीज़मैन इंस्टीट्यूट के विशेषज्ञों ने अणुओं और एंजाइमों से युक्त एक प्रोग्राम योग्य आणविक कंप्यूटिंग मशीन के निर्माण की घोषणा की थी। उन्होंने सिलिकॉन माइक्रोचिप्स को उनके साथ बदल दिया। आज तक, टीम ने और प्रगति की है। अब केवल एक डीएनए अणु एक कंप्यूटर को आवश्यक डेटा और आवश्यक ईंधन प्रदान कर सकता है।
जैव रासायनिक "नैनो कंप्यूटर" कोई कल्पना नहीं है; वे पहले से ही प्रकृति में मौजूद हैं और हर जीवित प्राणी में प्रकट होते हैं। लेकिन अक्सर इन्हें लोगों द्वारा प्रबंधित नहीं किया जाता है। कोई व्यक्ति "पाई" संख्या की गणना करने के लिए अभी तक किसी भी पौधे के जीनोम पर काम नहीं कर सकता है।
डेटा भंडारण/प्रसंस्करण के लिए डीएनए का उपयोग करने का विचार पहली बार 1994 में वैज्ञानिकों के दिमाग में आया था। यह तब था जब एक सरल गणितीय समस्या को हल करने के लिए एक अणु का उपयोग किया गया था। तब से, कई शोध समूहों ने डीएनए कंप्यूटर से संबंधित विभिन्न परियोजनाओं का प्रस्ताव दिया है। लेकिन यहां सभी प्रयास केवल ऊर्जा अणु पर आधारित थे। ऐसे कंप्यूटर को आप नंगी आंखों से नहीं देख सकते, यह टेस्ट ट्यूब में पानी के पारदर्शी घोल जैसा दिखता है। इसमें कोई यांत्रिक भाग नहीं हैं, बल्कि केवल खरबों जैव-आणविक उपकरण हैं - और यह केवल तरल की एक बूंद में है!
मानव डीएनए
लोग मानव डीएनए के प्रकार के बारे में 1953 में जागरूक हुए, जब वैज्ञानिक पहली बार दुनिया को डबल-स्ट्रैंडेड डीएनए मॉडल प्रदर्शित करने में सक्षम हुए। इसके लिए किर्क और वॉटसन को नोबेल पुरस्कार मिला, क्योंकि यह खोज 20वीं सदी में मौलिक हो गई थी।
समय के साथ, निश्चित रूप से, उन्होंने साबित कर दिया कि एक संरचित मानव अणु न केवल प्रस्तावित संस्करण जैसा दिख सकता है। अधिक विस्तृत डीएनए विश्लेषण करने के बाद, उन्होंने ए-, बी- और बाएं हाथ के फॉर्म जेड- की खोज की। फॉर्म ए- अक्सर एक अपवाद होता है, क्योंकि यह नमी की कमी होने पर ही बनता है। लेकिन यह केवल प्रयोगशाला अध्ययनों में ही संभव है; प्राकृतिक वातावरण के लिए यह असामान्य है; ऐसी प्रक्रिया जीवित कोशिका में नहीं हो सकती है।
बी-आकार क्लासिक है और इसे डबल दाएं हाथ की श्रृंखला के रूप में जाना जाता है, लेकिन जेड-आकार न केवल बाईं ओर विपरीत दिशा में मुड़ा हुआ है, बल्कि इसमें अधिक ज़िगज़ैग उपस्थिति भी है। वैज्ञानिकों ने जी-क्वाड्रुप्लेक्स रूप की भी पहचान कर ली है। इसकी संरचना में 2 नहीं, बल्कि 4 धागे हैं। आनुवंशिकीविदों के अनुसार यह रूप उन क्षेत्रों में होता है जहां ग्वानिन की अधिक मात्रा होती है।

कृत्रिम डीएनए
आज पहले से ही कृत्रिम डीएनए मौजूद है, जो असली की एक समान प्रतिलिपि है; यह पूरी तरह से प्राकृतिक डबल हेलिक्स की संरचना का अनुसरण करता है। लेकिन, मूल पॉलीन्यूक्लियोटाइड के विपरीत, कृत्रिम में केवल दो अतिरिक्त न्यूक्लियोटाइड होते हैं।
चूंकि डबिंग वास्तविक डीएनए के विभिन्न अध्ययनों से प्राप्त जानकारी के आधार पर बनाई गई थी, इसलिए इसे कॉपी, स्व-प्रतिकृति और विकसित भी किया जा सकता है। विशेषज्ञ ऐसे कृत्रिम अणु के निर्माण पर लगभग 20 वर्षों से काम कर रहे हैं। परिणाम एक अद्भुत आविष्कार है जो प्राकृतिक डीएनए की तरह ही आनुवंशिक कोड का उपयोग कर सकता है।
चार मौजूदा नाइट्रोजनस आधारों में, आनुवंशिकीविदों ने दो अतिरिक्त आधार जोड़े, जो प्राकृतिक आधारों के रासायनिक संशोधन द्वारा बनाए गए थे। प्राकृतिक डीएनए के विपरीत, कृत्रिम डीएनए काफी छोटा निकला। इसमें केवल 81 आधार जोड़े हैं। हालाँकि, यह प्रजनन और विकास भी करता है।
कृत्रिम रूप से प्राप्त अणु की प्रतिकृति पोलीमरेज़ श्रृंखला प्रतिक्रिया के कारण होती है, लेकिन अभी तक यह स्वतंत्र रूप से नहीं, बल्कि वैज्ञानिकों के हस्तक्षेप से होता है। वे स्वतंत्र रूप से उक्त डीएनए में आवश्यक एंजाइम जोड़ते हैं, इसे विशेष रूप से तैयार तरल माध्यम में रखते हैं।
अंतिम परिणाम
डीएनए विकास की प्रक्रिया और अंतिम परिणाम उत्परिवर्तन जैसे विभिन्न कारकों से प्रभावित हो सकते हैं। इससे पदार्थ के नमूनों का अध्ययन करना आवश्यक हो जाता है ताकि विश्लेषण परिणाम विश्वसनीय और विश्वसनीय हो। एक उदाहरण पितृत्व परीक्षण है. लेकिन हम इस बात से खुश हुए बिना नहीं रह सकते कि उत्परिवर्तन जैसी घटनाएं दुर्लभ हैं। फिर भी, विश्लेषण के आधार पर अधिक सटीक जानकारी प्राप्त करने के लिए पदार्थ के नमूनों की हमेशा दोबारा जाँच की जाती है।

प्लांट डीएनए
उच्च अनुक्रमण प्रौद्योगिकियों (एचटीएस) के लिए धन्यवाद, जीनोमिक्स के क्षेत्र में एक क्रांति हुई है - पौधों से डीएनए निष्कर्षण भी संभव है। बेशक, पौधों की सामग्री से उच्च गुणवत्ता वाले आणविक भार डीएनए प्राप्त करने में माइटोकॉन्ड्रिया और क्लोरोप्लास्ट डीएनए की बड़ी संख्या के साथ-साथ पॉलीसेकेराइड और फेनोलिक यौगिकों के उच्च स्तर के कारण कुछ कठिनाइयां पैदा होती हैं। इस मामले में हम जिस संरचना पर विचार कर रहे हैं उसे अलग करने के लिए विभिन्न तरीकों का उपयोग किया जाता है।
डीएनए में हाइड्रोजन बंधन
डीएनए अणु में हाइड्रोजन बंधन एक धनात्मक आवेशित हाइड्रोजन परमाणु के बीच उत्पन्न विद्युत चुम्बकीय आकर्षण के लिए जिम्मेदार होता है जो एक विद्युत ऋणात्मक परमाणु से जुड़ा होता है। यह द्विध्रुवीय अंतःक्रिया रासायनिक बंधन की कसौटी पर खरी नहीं उतरती। लेकिन यह अंतरआण्विक रूप से या अणु के विभिन्न भागों में, यानी इंट्रामोल्युलर रूप से हो सकता है।

एक हाइड्रोजन परमाणु विद्युत ऋणात्मक परमाणु से जुड़ता है जो बंधन का दाता है। एक विद्युत ऋणात्मक परमाणु नाइट्रोजन, फ्लोरीन या ऑक्सीजन हो सकता है। यह - विकेंद्रीकरण के माध्यम से - हाइड्रोजन नाभिक से इलेक्ट्रॉन बादल को अपनी ओर आकर्षित करता है और हाइड्रोजन परमाणु (आंशिक रूप से) को सकारात्मक रूप से चार्ज करता है। चूँकि H का आकार अन्य अणुओं और परमाणुओं की तुलना में छोटा है, इसलिए आवेश भी छोटा है।
डीएनए डिकोडिंग
डीएनए अणु को समझने से पहले, वैज्ञानिक पहले बड़ी संख्या में कोशिकाएं लेते हैं। सबसे सटीक और सफल कार्य के लिए, उनमें से लगभग दस लाख की आवश्यकता होती है। अध्ययन के दौरान प्राप्त परिणामों की लगातार तुलना की जाती है और उन्हें रिकॉर्ड किया जाता है। आज, जीनोम डिकोडिंग अब दुर्लभ नहीं है, बल्कि एक सुलभ प्रक्रिया है।

बेशक, किसी एकल कोशिका के जीनोम को समझना एक अव्यावहारिक अभ्यास है। ऐसे अध्ययनों के दौरान प्राप्त आंकड़ों में वैज्ञानिकों की कोई दिलचस्पी नहीं है। लेकिन यह समझना महत्वपूर्ण है कि वर्तमान में मौजूद सभी डिकोडिंग विधियां, उनकी जटिलता के बावजूद, पर्याप्त प्रभावी नहीं हैं। वे डीएनए का केवल 40-70% ही पढ़ने की अनुमति देंगे।
हालाँकि, हार्वर्ड के प्रोफेसरों ने हाल ही में एक ऐसी विधि की घोषणा की जिसके माध्यम से 90% जीनोम को समझा जा सकता है। यह तकनीक पृथक कोशिकाओं में प्राइमर अणुओं को जोड़ने पर आधारित है, जिसकी मदद से डीएनए प्रतिकृति शुरू होती है। लेकिन फिर भी इस पद्धति को सफल नहीं माना जा सकता; विज्ञान में खुले तौर पर उपयोग करने से पहले इसे अभी भी परिष्कृत करने की आवश्यकता है।
न्यूक्लिक एसिड मोनोन्यूक्लियोटाइड्स से युक्त उच्च-आणविक पदार्थ होते हैं, जो 3", 5" फॉस्फोडिएस्टर बांड का उपयोग करके एक बहुलक श्रृंखला में एक दूसरे से जुड़े होते हैं और एक निश्चित तरीके से कोशिकाओं में पैक किए जाते हैं।
न्यूक्लिक एसिड दो प्रकार के बायोपॉलिमर होते हैं: राइबोन्यूक्लिक एसिड (आरएनए) और डीऑक्सीराइबोन्यूक्लिक एसिड (डीएनए)। प्रत्येक बायोपॉलिमर में न्यूक्लियोटाइड होते हैं जो कार्बोहाइड्रेट अवशेषों (राइबोस, डीऑक्सीराइबोज) और नाइट्रोजनस बेस (यूरैसिल, थाइमिन) में से एक में भिन्न होते हैं। इन अंतरों के अनुसार, न्यूक्लिक एसिड को उनका नाम मिला।
डीऑक्सीराइबोन्यूक्लिक एसिड की संरचना
न्यूक्लिक एसिड की प्राथमिक, द्वितीयक और तृतीयक संरचना होती है।
डीएनए की प्राथमिक संरचना
डीएनए की प्राथमिक संरचना एक रैखिक पॉलीन्यूक्लियोटाइड श्रृंखला है जिसमें मोनोन्यूक्लियोटाइड्स 3", 5" फॉस्फोडिएस्टर बांड द्वारा जुड़े होते हैं। एक कोशिका में न्यूक्लिक एसिड श्रृंखला के संयोजन के लिए प्रारंभिक सामग्री 5"-ट्राइफॉस्फेट न्यूक्लियोसाइड है, जो β और γ फॉस्फोरिक एसिड अवशेषों को हटाने के परिणामस्वरूप, दूसरे न्यूक्लियोसाइड के 3" कार्बन परमाणु को जोड़ने में सक्षम है। . इस प्रकार, एक डीऑक्सीराइबोज़ का 3" कार्बन परमाणु एकल फॉस्फोरिक एसिड अवशेष के माध्यम से दूसरे डीऑक्सीराइबोज़ के 5" कार्बन परमाणु से सहसंयोजक रूप से जुड़ा होता है और न्यूक्लिक एसिड की एक रैखिक पॉलीन्यूक्लियोटाइड श्रृंखला बनाता है। इसलिए नाम: 3", 5" फॉस्फोडाइस्टर बांड। नाइट्रोजन आधार एक श्रृंखला के न्यूक्लियोटाइड को जोड़ने में भाग नहीं लेते हैं (चित्र 1.)।
एक न्यूक्लियोटाइड के फॉस्फोरिक एसिड अणु अवशेष और दूसरे के कार्बोहाइड्रेट के बीच ऐसा संबंध, पॉलीन्यूक्लियोटाइड अणु के एक पेंटोस-फॉस्फेट कंकाल के गठन की ओर जाता है, जिस पर नाइट्रोजनस आधार एक के बाद एक जुड़े होते हैं। न्यूक्लिक एसिड अणुओं की श्रृंखलाओं में उनकी व्यवस्था का क्रम विभिन्न जीवों की कोशिकाओं के लिए सख्ती से विशिष्ट है, अर्थात। एक विशिष्ट चरित्र है (शार्गफ का नियम)।
एक रैखिक डीएनए श्रृंखला, जिसकी लंबाई श्रृंखला में शामिल न्यूक्लियोटाइड की संख्या पर निर्भर करती है, के दो सिरे होते हैं: एक को 3" सिरा कहा जाता है और इसमें एक मुक्त हाइड्रॉक्सिल होता है, और दूसरे को 5" सिरा कहा जाता है और इसमें फॉस्फोरिक होता है। अम्ल अवशेष. सर्किट ध्रुवीय है और इसकी दिशा 5"->3" और 3"->5" हो सकती है। अपवाद गोलाकार डीएनए है।
डीएनए का आनुवंशिक "पाठ" कोड "शब्दों" से बना है - न्यूक्लियोटाइड के त्रिक जिन्हें कोडन कहा जाता है। सभी प्रकार के आरएनए की प्राथमिक संरचना के बारे में जानकारी रखने वाले डीएनए के अनुभागों को संरचनात्मक जीन कहा जाता है।
पॉलीन्यूक्लियोटाइड डीएनए श्रृंखलाएं विशाल आकार तक पहुंचती हैं, इसलिए वे कोशिका में एक निश्चित तरीके से पैक की जाती हैं।
डीएनए की संरचना का अध्ययन करते समय, चारगफ़ (1949) ने व्यक्तिगत डीएनए आधारों की सामग्री के संबंध में महत्वपूर्ण पैटर्न स्थापित किए। उन्होंने डीएनए की द्वितीयक संरचना को प्रकट करने में मदद की। इन पैटर्नों को चारगफ़ के नियम कहा जाता है। चारगफ़ नियम
इन नियमों से संकेत मिलता है कि डीएनए का निर्माण करते समय, सामान्य रूप से प्यूरीन और पाइरीमिडीन आधारों का नहीं, बल्कि विशेष रूप से एडेनिन के साथ थाइमिन और गुआनिन के साथ साइटोसिन का काफी सख्त पत्राचार (युग्मन) देखा जाना चाहिए। इन नियमों के आधार पर, 1953 में, वॉटसन और क्रिक ने डीएनए की द्वितीयक संरचना का एक मॉडल प्रस्तावित किया, जिसे डबल हेलिक्स (चित्र) कहा जाता है। |

डीएनए की द्वितीयक संरचना
डीएनए की द्वितीयक संरचना एक डबल हेलिक्स है, जिसका मॉडल 1953 में डी. वाटसन और एफ. क्रिक द्वारा प्रस्तावित किया गया था।
डीएनए मॉडल बनाने के लिए पूर्वापेक्षाएँ
प्रारंभिक विश्लेषणों के परिणामस्वरूप, यह माना गया कि किसी भी मूल के डीएनए में सभी चार न्यूक्लियोटाइड समान दाढ़ मात्रा में होते हैं। हालाँकि, 1940 के दशक में, ई. चारगफ़ और उनके सहयोगियों ने, विभिन्न प्रकार के जीवों से पृथक डीएनए का विश्लेषण करने के परिणामस्वरूप, स्पष्ट रूप से दिखाया कि उनमें विभिन्न मात्रात्मक अनुपात में नाइट्रोजनस आधार मौजूद थे। चारगफ़ ने पाया कि यद्यपि ये अनुपात जीवों की एक ही प्रजाति की सभी कोशिकाओं के डीएनए के लिए समान हैं, विभिन्न प्रजातियों के डीएनए कुछ न्यूक्लियोटाइड की सामग्री में स्पष्ट रूप से भिन्न हो सकते हैं। इससे पता चला कि नाइट्रोजनस आधारों के अनुपात में अंतर किसी प्रकार के जैविक कोड से जुड़ा हो सकता है। यद्यपि विभिन्न डीएनए नमूनों में व्यक्तिगत प्यूरीन और पाइरीमिडीन आधारों का अनुपात अलग-अलग निकला, परीक्षण के परिणामों की तुलना करने पर, एक निश्चित पैटर्न सामने आया: सभी नमूनों में, प्यूरीन की कुल संख्या पाइरीमिडीन की कुल संख्या के बराबर थी (ए + जी = टी + सी), एडेनिन की मात्रा थाइमिन (ए = टी) की मात्रा के बराबर थी, और ग्वानिन की मात्रा साइटोसिन (जी = सी) की मात्रा के बराबर थी। स्तनधारी कोशिकाओं से अलग किए गए डीएनए में आमतौर पर एडेनिन और थाइमिन की मात्रा अधिक होती है और गुआनिन और साइटोसिन की मात्रा अपेक्षाकृत कम होती है, जबकि बैक्टीरिया के डीएनए में गुआनिन और साइटोसिन की मात्रा अधिक होती है और एडेनिन और थाइमिन की मात्रा अपेक्षाकृत कम होती है। इन आंकड़ों ने तथ्यात्मक सामग्री का एक महत्वपूर्ण हिस्सा बनाया जिसके आधार पर बाद में डीएनए संरचना का वाटसन-क्रिक मॉडल बनाया गया।
डीएनए की संभावित संरचना का एक और महत्वपूर्ण अप्रत्यक्ष संकेत प्रोटीन अणुओं की संरचना पर एल. पॉलिंग के डेटा द्वारा प्रदान किया गया था। पॉलिंग ने दिखाया कि प्रोटीन अणु में अमीनो एसिड श्रृंखला के कई अलग-अलग स्थिर विन्यास संभव हैं। एक सामान्य पेप्टाइड श्रृंखला विन्यास, α-हेलिक्स, एक नियमित पेचदार संरचना है। इस संरचना के साथ, श्रृंखला के आसन्न घुमावों पर स्थित अमीनो एसिड के बीच हाइड्रोजन बांड का निर्माण संभव है। पॉलिंग ने 1950 में पॉलीपेप्टाइड श्रृंखला के α-पेचदार विन्यास का वर्णन किया और सुझाव दिया कि डीएनए अणुओं में संभवतः हाइड्रोजन बांड द्वारा जगह पर एक पेचदार संरचना होती है।
हालाँकि, डीएनए अणु की संरचना के बारे में सबसे मूल्यवान जानकारी एक्स-रे विवर्तन विश्लेषण के परिणामों द्वारा प्रदान की गई थी। डीएनए क्रिस्टल से गुजरने वाली एक्स-रे विवर्तन से गुजरती हैं, यानी वे कुछ दिशाओं में विक्षेपित हो जाती हैं। किरणों के विक्षेपण की डिग्री और प्रकृति स्वयं अणुओं की संरचना पर निर्भर करती है। एक एक्स-रे विवर्तन पैटर्न (चित्र 3) अनुभवी आंख को अध्ययन के तहत पदार्थ के अणुओं की संरचना के संबंध में कई अप्रत्यक्ष संकेत देता है। डीएनए के एक्स-रे विवर्तन पैटर्न के विश्लेषण से यह निष्कर्ष निकला कि नाइट्रोजनस आधार (जिनका आकार सपाट होता है) प्लेटों के ढेर की तरह व्यवस्थित होते हैं। एक्स-रे विवर्तन पैटर्न से क्रिस्टलीय डीएनए की संरचना में तीन मुख्य अवधियों का पता चला: 0.34, 2 और 3.4 एनएम।

वॉटसन-क्रिक डीएनए मॉडल
चारगफ के विश्लेषणात्मक डेटा, विल्किंस के एक्स-रे पैटर्न और रसायनज्ञों के शोध के आधार पर, जिन्होंने एक अणु में परमाणुओं के बीच की सटीक दूरी, किसी दिए गए परमाणु के बंधन के बीच के कोण और परमाणुओं के आकार के बारे में जानकारी प्रदान की, वाटसन और क्रिक ने एक निश्चित पैमाने पर डीएनए अणु के व्यक्तिगत घटकों के भौतिक मॉडल बनाना शुरू किया और उन्हें एक-दूसरे के साथ इस तरह से "समायोजित" किया कि परिणामी प्रणाली विभिन्न प्रयोगात्मक डेटा से मेल खाती है। [दिखाओ] .
यह पहले भी ज्ञात था कि डीएनए श्रृंखला में पड़ोसी न्यूक्लियोटाइड फॉस्फोडिएस्टर पुलों से जुड़े होते हैं, जो एक न्यूक्लियोटाइड के 5"-कार्बन डीऑक्सीराइबोज परमाणु को अगले न्यूक्लियोटाइड के 3"-कार्बन डीऑक्सीराइबोज परमाणु से जोड़ते हैं। वॉटसन और क्रिक को इसमें कोई संदेह नहीं था कि 0.34 एनएम की अवधि डीएनए श्रृंखला में क्रमिक न्यूक्लियोटाइड के बीच की दूरी से मेल खाती है। इसके अलावा, यह माना जा सकता है कि 2 एनएम की अवधि श्रृंखला की मोटाई से मेल खाती है। और यह समझाने के लिए कि 3.4 एनएम की अवधि किस वास्तविक संरचना से मेल खाती है, वॉटसन और क्रिक, साथ ही पॉलिंग ने पहले सुझाव दिया था कि श्रृंखला एक सर्पिल के रूप में मुड़ जाती है (या, अधिक सटीक रूप से, एक पेचदार रेखा बनाती है, क्योंकि इन शब्दों के सख्त अर्थ में एक सर्पिल तब प्राप्त होता है जब कुंडलियाँ अंतरिक्ष में बेलनाकार सतह के बजाय शंक्वाकार सतह बनाती हैं)। फिर 3.4 एनएम की अवधि इस हेलिक्स के क्रमिक घुमावों के बीच की दूरी के अनुरूप होगी। ऐसा सर्पिल बहुत घना या कुछ हद तक फैला हुआ हो सकता है, यानी इसके मोड़ सपाट या खड़े हो सकते हैं। चूँकि 3.4 एनएम की अवधि क्रमिक न्यूक्लियोटाइड्स (0.34 एनएम) के बीच की दूरी का ठीक 10 गुना है, इसलिए यह स्पष्ट है कि हेलिक्स के प्रत्येक पूर्ण मोड़ में 10 न्यूक्लियोटाइड होते हैं। इन आंकड़ों से, वॉटसन और क्रिक 2 एनएम के व्यास के साथ एक हेलिक्स में मुड़ी हुई पॉलीन्यूक्लियोटाइड श्रृंखला के घनत्व की गणना करने में सक्षम थे, जिसमें 3.4 एनएम के घुमावों के बीच की दूरी थी। यह पता चला कि ऐसी श्रृंखला का घनत्व डीएनए के वास्तविक घनत्व का आधा होगा, जो पहले से ही ज्ञात था। मुझे यह मानना पड़ा कि डीएनए अणु में दो श्रृंखलाएं होती हैं - कि यह न्यूक्लियोटाइड्स का एक डबल हेलिक्स है।
निस्संदेह, अगला कार्य डबल हेलिक्स बनाने वाली दो श्रृंखलाओं के बीच स्थानिक संबंधों को स्पष्ट करना था। अपने भौतिक मॉडल पर श्रृंखलाओं की व्यवस्था के लिए कई विकल्पों की कोशिश करने के बाद, वॉटसन और क्रिक ने पाया कि सभी उपलब्ध डेटा उस विकल्प से सबसे अच्छे से मेल खाते थे जिसमें दो पॉलीन्यूक्लियोटाइड हेलिकॉप्टर विपरीत दिशाओं में जाते हैं; इस मामले में, चीनी और फॉस्फेट अवशेषों से युक्त श्रृंखलाएं डबल हेलिक्स की सतह बनाती हैं, और प्यूरीन और पाइरीमिडीन अंदर स्थित होते हैं। एक दूसरे के विपरीत स्थित आधार, दो श्रृंखलाओं से संबंधित, हाइड्रोजन बांड द्वारा जोड़े में जुड़े हुए हैं; ये हाइड्रोजन बांड हैं जो श्रृंखलाओं को एक साथ रखते हैं, इस प्रकार अणु के समग्र विन्यास को ठीक करते हैं।
डीएनए के दोहरे हेलिक्स की कल्पना एक रस्सी की सीढ़ी के रूप में की जा सकती है जिसे पेचदार तरीके से घुमाया जाता है, ताकि इसके पायदान क्षैतिज रहें। फिर दो अनुदैर्ध्य रस्सियाँ चीनी और फॉस्फेट अवशेषों की श्रृंखलाओं के अनुरूप होंगी, और क्रॉसबार हाइड्रोजन बांड से जुड़े नाइट्रोजनस आधारों के जोड़े के अनुरूप होंगे।
संभावित मॉडलों के आगे के अध्ययन के परिणामस्वरूप, वॉटसन और क्रिक ने निष्कर्ष निकाला कि प्रत्येक "क्रॉसबार" में एक प्यूरीन और एक पाइरीमिडीन शामिल होना चाहिए; 2 एनएम (डबल हेलिक्स के व्यास के अनुरूप) की अवधि में, दो प्यूरीन के लिए पर्याप्त जगह नहीं होगी, और दो पाइरीमिडीन उचित हाइड्रोजन बांड बनाने के लिए एक दूसरे के करीब नहीं हो सकते हैं। विस्तृत मॉडल के गहन अध्ययन से पता चला कि एडेनिन और साइटोसिन, एक उपयुक्त आकार का संयोजन बनाते समय, अभी भी इस तरह से स्थित नहीं हो सके हैं कि उनके बीच हाइड्रोजन बांड बन सकें। इसी तरह की रिपोर्टों ने गुआनिन - थाइमिन के संयोजन को बाहर करने के लिए मजबूर किया, जबकि एडेनिन - थाइमिन और गुआनिन - साइटोसिन के संयोजन काफी स्वीकार्य निकले। हाइड्रोजन बंधों की प्रकृति ऐसी होती है कि एडेनिन थाइमिन के साथ और गुआनिन साइटोसिन के साथ जोड़ी बनाता है। विशिष्ट आधार युग्मन के इस विचार ने "चारगफ़ नियम" की व्याख्या करना संभव बना दिया, जिसके अनुसार किसी भी डीएनए अणु में एडेनिन की मात्रा हमेशा थाइमिन की सामग्री के बराबर होती है, और गुआनिन की मात्रा हमेशा मात्रा के बराबर होती है साइटोसिन का. एडेनिन और थाइमिन के बीच दो हाइड्रोजन बांड बनते हैं, और गुआनिन और साइटोसिन के बीच तीन हाइड्रोजन बांड बनते हैं। इस विशिष्टता के कारण, एक श्रृंखला में प्रत्येक एडेनिन के खिलाफ हाइड्रोजन बांड के गठन से दूसरे पर थाइमिन बनता है; उसी प्रकार, केवल साइटोसिन ही प्रत्येक गुआनिन के विपरीत हो सकता है। इस प्रकार, शृंखलाएँ एक-दूसरे की पूरक हैं, अर्थात्, एक शृंखला में न्यूक्लियोटाइड का क्रम दूसरे में उनके अनुक्रम को विशिष्ट रूप से निर्धारित करता है। दो श्रृंखलाएँ विपरीत दिशाओं में चलती हैं और उनके टर्मिनल फॉस्फेट समूह डबल हेलिक्स के विपरीत छोर पर हैं।
अपने शोध के परिणामस्वरूप, 1953 में वॉटसन और क्रिक ने डीएनए अणु की संरचना का एक मॉडल प्रस्तावित किया (चित्र 3), जो आज भी प्रासंगिक है। मॉडल के अनुसार, डीएनए अणु में दो पूरक पॉलीन्यूक्लियोटाइड श्रृंखलाएं होती हैं। प्रत्येक डीएनए स्ट्रैंड एक पॉलीन्यूक्लियोटाइड है जिसमें कई दसियों हज़ार न्यूक्लियोटाइड होते हैं। इसमें, पड़ोसी न्यूक्लियोटाइड एक मजबूत सहसंयोजक बंधन द्वारा फॉस्फोरिक एसिड अवशेष और डीऑक्सीराइबोज के कनेक्शन के कारण एक नियमित पेंटोस-फॉस्फेट रीढ़ बनाते हैं। एक पॉलीन्यूक्लियोटाइड श्रृंखला के नाइट्रोजनस आधारों को दूसरे के नाइट्रोजनस आधारों के विपरीत कड़ाई से परिभाषित क्रम में व्यवस्थित किया जाता है। पॉलीन्यूक्लियोटाइड श्रृंखला में नाइट्रोजनस आधारों का प्रत्यावर्तन अनियमित है।
डीएनए श्रृंखला में नाइट्रोजनस आधारों की व्यवस्था पूरक है (ग्रीक "पूरक" से - जोड़), यानी। थाइमिन (टी) हमेशा एडेनिन (ए) के खिलाफ होता है, और केवल साइटोसिन (सी) गुआनिन (जी) के खिलाफ होता है। यह इस तथ्य से समझाया गया है कि ए और टी, साथ ही जी और सी, एक-दूसरे से सख्ती से मेल खाते हैं, यानी। एक दूसरे की पूर्ति करना। यह पत्राचार आधारों की रासायनिक संरचना द्वारा निर्धारित किया जाता है, जो प्यूरीन और पाइरीमिडीन जोड़ी में हाइड्रोजन बांड के गठन की अनुमति देता है। A और T के बीच दो कनेक्शन हैं, और G और C के बीच तीन कनेक्शन हैं। ये बंधन अंतरिक्ष में डीएनए अणु का आंशिक स्थिरीकरण प्रदान करते हैं। डबल हेलिक्स की स्थिरता सीधे G≡C बांड की संख्या के समानुपाती होती है, जो A=T बांड की तुलना में अधिक स्थिर होती है।
एक डीएनए श्रृंखला में न्यूक्लियोटाइड की व्यवस्था का ज्ञात अनुक्रम, पूरकता के सिद्धांत के अनुसार, दूसरी श्रृंखला के न्यूक्लियोटाइड को स्थापित करना संभव बनाता है।
इसके अलावा, यह स्थापित किया गया है कि जलीय घोल में सुगंधित संरचना वाले नाइट्रोजनस आधार एक के ऊपर एक स्थित होते हैं, जिससे सिक्कों का ढेर बनता है। कार्बनिक अणुओं के ढेर बनाने की इस प्रक्रिया को स्टैकिंग कहा जाता है। विचाराधीन वॉटसन-क्रिक मॉडल के डीएनए अणु की पॉलीन्यूक्लियोटाइड श्रृंखलाओं में एक समान भौतिक-रासायनिक अवस्था होती है, उनके नाइट्रोजनस आधार सिक्कों के ढेर के रूप में व्यवस्थित होते हैं, जिनके विमानों के बीच वैन डेर वाल्स इंटरैक्शन (स्टैकिंग इंटरैक्शन) उत्पन्न होते हैं।
पूरक आधारों (क्षैतिज रूप से) के बीच हाइड्रोजन बंधन और वैन डेर वाल्स बलों (लंबवत) के कारण पॉलीन्यूक्लियोटाइड श्रृंखला में आधारों के विमानों के बीच स्टैकिंग इंटरैक्शन डीएनए अणु को अंतरिक्ष में अतिरिक्त स्थिरीकरण प्रदान करते हैं।
दोनों श्रृंखलाओं की चीनी फॉस्फेट रीढ़ की हड्डी बाहर की ओर होती है, और आधार एक दूसरे की ओर अंदर की ओर होते हैं। डीएनए में श्रृंखलाओं की दिशा प्रतिसमानांतर होती है (उनमें से एक की दिशा 5"->3", दूसरी की - 3"->5" होती है, अर्थात एक श्रृंखला का 3" सिरा 5" सिरे के विपरीत स्थित होता है) अन्य।)। जंजीरें एक सामान्य अक्ष के साथ दाएं हाथ के सर्पिल बनाती हैं। हेलिक्स का एक मोड़ 10 न्यूक्लियोटाइड है, मोड़ का आकार 3.4 एनएम है, प्रत्येक न्यूक्लियोटाइड की ऊंचाई 0.34 एनएम है, हेलिक्स का व्यास 2.0 एनएम है। एक स्ट्रैंड के दूसरे के चारों ओर घूमने के परिणामस्वरूप, डीएनए डबल हेलिक्स की एक बड़ी नाली (लगभग 20 Å व्यास) और एक छोटी नाली (लगभग 12 Å व्यास) बनती है। वॉटसन-क्रिक डबल हेलिक्स के इस रूप को बाद में बी-फॉर्म कहा गया। कोशिकाओं में, डीएनए आमतौर पर बी रूप में मौजूद होता है, जो सबसे अधिक स्थिर होता है।
डीएनए के कार्य
प्रस्तावित मॉडल ने डीऑक्सीराइबोन्यूक्लिक एसिड के कई जैविक गुणों की व्याख्या की, जिसमें आनुवंशिक जानकारी का भंडारण और 4 न्यूक्लियोटाइड के अनुक्रमिक संयोजनों की एक विस्तृत विविधता द्वारा प्रदान की गई जीन की विविधता और एक आनुवंशिक कोड के अस्तित्व का तथ्य, स्व-प्रजनन की क्षमता शामिल है। और प्रतिकृति प्रक्रिया द्वारा प्रदान की गई आनुवंशिक जानकारी को प्रसारित करना, और प्रोटीन के रूप में आनुवंशिक जानकारी के कार्यान्वयन के साथ-साथ एंजाइम प्रोटीन की मदद से बनने वाले किसी भी अन्य यौगिक को प्रसारित करना।
डीएनए के बुनियादी कार्य.
- डीएनए आनुवंशिक जानकारी का वाहक है, जो आनुवंशिक कोड के अस्तित्व से सुनिश्चित होता है।
- कोशिकाओं और जीवों की पीढ़ियों में आनुवंशिक जानकारी का पुनरुत्पादन और संचरण। यह कार्यक्षमता प्रतिकृति प्रक्रिया द्वारा प्रदान की जाती है।
- प्रोटीन के साथ-साथ एंजाइम प्रोटीन की मदद से बनने वाले किसी भी अन्य यौगिक के रूप में आनुवंशिक जानकारी का कार्यान्वयन। यह फ़ंक्शन प्रतिलेखन और अनुवाद की प्रक्रियाओं द्वारा प्रदान किया जाता है।

डबल-स्ट्रैंडेड डीएनए के संगठन के रूप
डीएनए कई प्रकार के डबल हेलिकॉप्टर बना सकता है (चित्र 4)। वर्तमान में, छह रूप पहले से ही ज्ञात हैं (ए से ई और जेड-फॉर्म तक)।
डीएनए के संरचनात्मक रूप, जैसा कि रोज़ालिंड फ्रैंकलिन ने स्थापित किया था, पानी के साथ न्यूक्लिक एसिड अणु की संतृप्ति पर निर्भर करता है। एक्स-रे विवर्तन विश्लेषण का उपयोग करके डीएनए फाइबर के अध्ययन में, यह दिखाया गया कि एक्स-रे पैटर्न मौलिक रूप से सापेक्ष आर्द्रता पर निर्भर करता है कि प्रयोग इस फाइबर की जल संतृप्ति की किस डिग्री पर होता है। यदि फाइबर पानी से पर्याप्त रूप से संतृप्त था, तो एक रेडियोग्राफ़ प्राप्त किया गया था। सूखने पर, एक पूरी तरह से अलग एक्स-रे पैटर्न दिखाई दिया, जो उच्च नमी वाले फाइबर के एक्स-रे पैटर्न से बहुत अलग था।
उच्च आर्द्रता वाले डीएनए अणु को बी-फॉर्म कहा जाता है. शारीरिक स्थितियों (कम नमक सांद्रता, जलयोजन की उच्च डिग्री) के तहत, डीएनए का प्रमुख संरचनात्मक प्रकार बी-फॉर्म (डबल-स्ट्रैंडेड डीएनए का मुख्य रूप - वाटसन-क्रिक मॉडल) है। ऐसे अणु की हेलिक्स पिच 3.4 एनएम है। प्रति मोड़ में 10 पूरक जोड़े "सिक्के" के मुड़े हुए ढेर के रूप में होते हैं - नाइट्रोजनस आधार। ढेर को ढेर के दो विरोधी "सिक्के" के बीच हाइड्रोजन बांड द्वारा एक साथ रखा जाता है, और दाएं हाथ के हेलिक्स में मुड़े हुए फॉस्फोडिएस्टर रीढ़ की हड्डी के दो रिबन द्वारा "घाव" किया जाता है। नाइट्रोजनस आधारों के तल हेलिक्स की धुरी के लंबवत होते हैं। आसन्न पूरक जोड़े एक दूसरे के सापेक्ष 36° घूमते हैं। हेलिक्स का व्यास 20Å है, जिसमें प्यूरीन न्यूक्लियोटाइड 12Å और पाइरीमिडीन न्यूक्लियोटाइड 8Å है।
कम आर्द्रता वाले डीएनए अणु को ए-फॉर्म कहा जाता है. ए-फॉर्म कम उच्च जलयोजन की स्थिति और Na + या K + आयनों की उच्च सामग्री पर बनता है। इस व्यापक दाएं हाथ की पेचदार संरचना में प्रति मोड़ 11 आधार जोड़े हैं। नाइट्रोजनस आधारों के तलों का हेलिक्स अक्ष की ओर अधिक झुकाव होता है; वे सामान्य से हेलिक्स अक्ष की ओर 20° तक विचलित हो जाते हैं। इसका तात्पर्य 5Å व्यास के साथ एक आंतरिक शून्य की उपस्थिति से है। आसन्न न्यूक्लियोटाइड के बीच की दूरी 0.23 एनएम है, मोड़ की लंबाई 2.5 एनएम है, और हेलिक्स का व्यास 2.3 एनएम है।
शुरुआत में डीएनए के ए रूप को कम महत्वपूर्ण माना गया था। हालाँकि, बाद में यह स्पष्ट हो गया कि डीएनए के ए-रूप का, बी-रूप की तरह, अत्यधिक जैविक महत्व है। टेम्प्लेट-प्राइमर कॉम्प्लेक्स में आरएनए-डीएनए हेलिक्स में ए-फॉर्म है, साथ ही आरएनए-आरएनए हेलिक्स और आरएनए हेयरपिन संरचनाएं हैं (राइबोस का 2'-हाइड्रॉक्सिल समूह आरएनए अणुओं को बी-फॉर्म बनाने से रोकता है)। डीएनए का ए-रूप बीजाणुओं में पाया जाता है। यह स्थापित किया गया है कि डीएनए का ए-रूप बी-रूप की तुलना में यूवी किरणों के प्रति 10 गुना अधिक प्रतिरोधी है।
ए-फॉर्म और बी-फॉर्म को डीएनए का कैनोनिकल फॉर्म कहा जाता है।
फॉर्म सी-ईदाएं हाथ के भी, उनका गठन केवल विशेष प्रयोगों में ही देखा जा सकता है, और, जाहिर है, वे विवो में मौजूद नहीं हैं। डीएनए के सी रूप की संरचना बी डीएनए के समान होती है। प्रति मोड़ आधार जोड़े की संख्या 9.33 है, हेलिक्स मोड़ की लंबाई 3.1 एनएम है। आधार जोड़े अक्ष की लंबवत स्थिति के सापेक्ष 8 डिग्री के कोण पर झुके हुए हैं। खांचे आकार में बी-डीएनए के खांचे के समान होते हैं। इस मामले में, मुख्य नाली कुछ उथली है, और छोटी नाली अधिक गहरी है। प्राकृतिक और सिंथेटिक डीएनए पॉलीन्यूक्लियोटाइड्स सी-फॉर्म में बदल सकते हैं।
| तालिका 1. कुछ प्रकार की डीएनए संरचनाओं के लक्षण | |||
| सर्पिल प्रकार | ए | बी | जेड |
| सर्पिल पिच | 0.32 एनएम | 3.38 एनएम | 4.46 एनएम |
| सर्पिल मोड़ | सही | सही | बाएं |
| प्रति मोड़ आधार जोड़े की संख्या | 11 | 10 | 12 |
| आधार तलों के बीच की दूरी | 0.256 एनएम | 0.338 एनएम | 0.371 एनएम |
| ग्लाइकोसिडिक बंधन संरचना | एंटी | एंटी | विरोधी ग गाओ |
| फ्यूरानोज़ रिंग की संरचना | C3"-एंडो | C2"-एंडो | C3"-एंडो-जी C2"-एंडो-सी |
| नाली की चौड़ाई, छोटी/बड़ी | 1.11/0.22 एनएम | 0.57/1.17 एनएम | 0.2/0.88 एनएम |
| नाली की गहराई, छोटी/बड़ी | 0.26/1.30 एनएम | 0.82/0.85 एनएम | 1.38/0.37 एनएम |
| सर्पिल व्यास | 2.3 एनएम | 2.0 एनएम | 1.8 एनएम |
डीएनए के संरचनात्मक तत्व
(गैर-विहित डीएनए संरचनाएं)
डीएनए के संरचनात्मक तत्वों में कुछ विशेष अनुक्रमों द्वारा सीमित असामान्य संरचनाएं शामिल हैं:
|

Z-आकार का डीएनएइसकी खोज 1979 में हेक्सान्यूक्लियोटाइड d(CG)3 - का अध्ययन करते समय की गई थी। इसकी खोज MIT के प्रोफेसर अलेक्जेंडर रिच और उनके सहयोगियों ने की थी। जेड-फॉर्म इस तथ्य के कारण डीएनए के सबसे महत्वपूर्ण संरचनात्मक तत्वों में से एक बन गया है कि इसका गठन डीएनए क्षेत्रों में देखा गया है जहां प्यूरीन पिरिमिडीन के साथ वैकल्पिक होते हैं (उदाहरण के लिए, 5'-जीसीजीसीजीसी-3'), या दोहराव 5 में '-CGCGCG-3' में मिथाइलेटेड साइटोसिन होता है। जेड-डीएनए के गठन और स्थिरीकरण के लिए एक आवश्यक शर्त सिन कन्फॉर्मेशन में इसमें प्यूरीन न्यूक्लियोटाइड्स की उपस्थिति थी, जो कि एंटी कन्फॉर्मेशन में पाइरीमिडीन बेस के साथ बारी-बारी से होती थी।
प्राकृतिक डीएनए अणु मुख्य रूप से दाएं हाथ के बी-रूप में मौजूद होते हैं जब तक कि उनमें (सीजी)एन जैसे अनुक्रम न हों। हालाँकि, यदि ऐसे अनुक्रम डीएनए का हिस्सा हैं, तो ये खंड, जब समाधान की आयनिक शक्ति या फॉस्फोडाइस्टर ढांचे पर नकारात्मक चार्ज को बेअसर करने वाले धनायन बदलते हैं, तो ये खंड जेड-फॉर्म में बदल सकते हैं, जबकि डीएनए के अन्य खंड श्रृंखला शास्त्रीय बी-रूप में रहती है। इस तरह के संक्रमण की संभावना इंगित करती है कि डीएनए डबल हेलिक्स में दो स्ट्रैंड एक गतिशील स्थिति में हैं और एक-दूसरे के सापेक्ष खुल सकते हैं, दाएं हाथ से बाएं हाथ की ओर जा सकते हैं और इसके विपरीत। ऐसी लैबिलिटी के जैविक परिणाम, जो डीएनए संरचना के गठनात्मक परिवर्तनों की अनुमति देते हैं, अभी तक पूरी तरह से समझ में नहीं आए हैं। ऐसा माना जाता है कि जेड-डीएनए के अनुभाग कुछ जीनों की अभिव्यक्ति को विनियमित करने में एक निश्चित भूमिका निभाते हैं और आनुवंशिक पुनर्संयोजन में भाग लेते हैं।
डीएनए का जेड-रूप एक बाएं हाथ का डबल हेलिक्स है जिसमें फॉस्फोडिएस्टर रीढ़ अणु की धुरी के साथ एक ज़िगज़ैग पैटर्न में स्थित है। इसलिए अणु का नाम (ज़िगज़ैग)-डीएनके है। ज़ेड-डीएनए प्रकृति में ज्ञात सबसे कम मुड़ा हुआ (प्रति मोड़ 12 आधार जोड़े) और सबसे पतला डीएनए है। आसन्न न्यूक्लियोटाइड के बीच की दूरी 0.38 एनएम है, मोड़ की लंबाई 4.56 एनएम है, और जेड-डीएनए का व्यास 1.8 एनएम है। इसके अलावा, इस डीएनए अणु की उपस्थिति एक एकल खांचे की उपस्थिति से भिन्न होती है।
डीएनए का Z रूप प्रोकैरियोटिक और यूकेरियोटिक कोशिकाओं में पाया गया है। अब ऐसे एंटीबॉडी प्राप्त हो गए हैं जो डीएनए के जेड-फॉर्म को बी-फॉर्म से अलग कर सकते हैं। ये एंटीबॉडीज़ ड्रोसोफिला (डॉ. मेलानोगास्टर) की लार ग्रंथि कोशिकाओं के विशाल गुणसूत्रों के कुछ क्षेत्रों से जुड़ते हैं। इन गुणसूत्रों की असामान्य संरचना के कारण बंधन प्रतिक्रिया की निगरानी करना आसान है, जिसमें सघन क्षेत्र (डिस्क) कम सघन क्षेत्रों (इंटरडिस्क) के विपरीत होते हैं। Z-डीएनए क्षेत्र इंटरडिस्क में स्थित हैं। इससे यह पता चलता है कि Z-फॉर्म वास्तव में प्राकृतिक परिस्थितियों में मौजूद है, हालांकि Z-फॉर्म के अलग-अलग वर्गों के आकार अभी भी अज्ञात हैं।

(इनवर्टर) डीएनए में सबसे प्रसिद्ध और अक्सर होने वाले आधार अनुक्रम हैं। पैलिंड्रोम एक शब्द या वाक्यांश है जो बाएँ से दाएँ और इसके विपरीत एक समान पढ़ता है। ऐसे शब्दों या वाक्यांशों के उदाहरण हैं: झोपड़ी, कोसैक, बाढ़, और अज़ोर के पंजे पर गिरा हुआ गुलाब। जब डीएनए अनुभागों पर लागू किया जाता है, तो इस शब्द (पैलिंड्रोम) का अर्थ श्रृंखला के साथ दाएं से बाएं और बाएं से दाएं (जैसे "हट" शब्द में अक्षर आदि) न्यूक्लियोटाइड का समान विकल्प होता है।
एक पैलिंड्रोम को आधार अनुक्रमों के उल्टे दोहराव की उपस्थिति की विशेषता है जिसमें दो डीएनए स्ट्रैंड के सापेक्ष दूसरे क्रम की समरूपता होती है। ऐसे अनुक्रम, स्पष्ट कारणों से, स्व-पूरक होते हैं और हेयरपिन या क्रूसिफ़ॉर्म संरचनाएं बनाते हैं (चित्र)। हेयरपिन नियामक प्रोटीन को यह पहचानने में मदद करते हैं कि गुणसूत्र डीएनए का आनुवंशिक पाठ कहाँ कॉपी किया गया है।
जब एक ही डीएनए स्ट्रैंड पर एक उलटा दोहराव मौजूद होता है, तो अनुक्रम को दर्पण दोहराव कहा जाता है। मिरर रिपीट में स्व-पूरक गुण नहीं होते हैं और इसलिए, हेयरपिन या क्रूसिफ़ॉर्म संरचनाएं बनाने में सक्षम नहीं होते हैं। इस प्रकार के अनुक्रम लगभग सभी बड़े डीएनए अणुओं में पाए जाते हैं और कुछ आधार जोड़े से लेकर कई हजार आधार जोड़े तक हो सकते हैं।
यूकेरियोटिक कोशिकाओं में क्रूसिफ़ॉर्म संरचनाओं के रूप में पैलिंड्रोम की उपस्थिति सिद्ध नहीं हुई है, हालांकि ई. कोली कोशिकाओं में क्रूसिफ़ॉर्म संरचनाओं की एक निश्चित संख्या का पता लगाया गया है। आरएनए या एकल-फंसे डीएनए में स्व-पूरक अनुक्रमों की उपस्थिति समाधान में न्यूक्लिक एसिड श्रृंखला को एक निश्चित स्थानिक संरचना में मोड़ने का मुख्य कारण है, जो कई "हेयरपिन" के गठन की विशेषता है।

एच-फॉर्म डीएनएतीन डीएनए स्ट्रैंड्स द्वारा निर्मित एक हेलिक्स है - एक डीएनए ट्रिपल हेलिक्स। यह वाटसन-क्रिक डबल हेलिक्स का एक कॉम्प्लेक्स है जिसमें तीसरा सिंगल-स्ट्रैंडेड डीएनए स्ट्रैंड होता है, जो इसके प्रमुख खांचे में फिट होता है, जिससे एक तथाकथित हुगस्टीन जोड़ी बनती है।
इस तरह के ट्रिपलेक्स का निर्माण डीएनए डबल हेलिक्स को इस तरह से मोड़ने के परिणामस्वरूप होता है कि इसका आधा भाग डबल हेलिक्स के रूप में रहता है, और दूसरा आधा अलग हो जाता है। इस मामले में, डिस्कनेक्ट किए गए हेलिकॉप्टरों में से एक डबल हेलिक्स के पहले आधे हिस्से के साथ एक नई संरचना बनाता है - एक ट्रिपल हेलिक्स, और दूसरा एकल-फंसे हुए खंड के रूप में, असंरचित हो जाता है। इस संरचनात्मक संक्रमण की एक विशेषता माध्यम के पीएच पर इसकी तीव्र निर्भरता है, जिसके प्रोटॉन नई संरचना को स्थिर करते हैं। इस विशेषता के कारण, नई संरचना को डीएनए का एच-फॉर्म कहा जाता था, जिसका गठन होमोप्यूरिन-होमोपाइरीमिडीन क्षेत्रों वाले सुपरकोइल्ड प्लास्मिड में खोजा गया था, जो एक दर्पण दोहराव है।
आगे के अध्ययनों में, यह स्थापित किया गया कि तीन-स्ट्रैंडेड संरचना के निर्माण के साथ कुछ होमोप्यूरिन-होमोपाइरीमिडीन डबल-स्ट्रैंडेड पॉलीन्यूक्लियोटाइड्स का संरचनात्मक संक्रमण करना संभव है:
- एक होमोप्यूरिन और दो होमोपाइरीमिडीन स्ट्रैंड ( पाय-पु-पाइ ट्रिपलक्स) [हुगस्टीन इंटरेक्शन]।
Py-Pu-Py ट्रिपलक्स के घटक ब्लॉक कैनोनिकल आइसोमोर्फिक CGC+ और TAT ट्रायड हैं। ट्रिपलक्स के स्थिरीकरण के लिए सीजीसी+ ट्रायड के प्रोटोनेशन की आवश्यकता होती है, इसलिए ये ट्रिपलक्स समाधान के पीएच पर निर्भर करते हैं।
- एक होमोपाइरीमिडीन और दो होमोप्यूरिन स्ट्रैंड ( पाय-पु-पु त्रिप्लेक्स) [उलटा हुगस्टीन इंटरैक्शन]।
Py-Pu-Pu ट्रिपलक्स के घटक ब्लॉक कैनोनिकल आइसोमोर्फिक CGG और TAA ट्रायड हैं। Py-Pu-Pu ट्रिपलेक्स की एक आवश्यक संपत्ति दोहरे चार्ज वाले आयनों की उपस्थिति पर उनकी स्थिरता की निर्भरता है, और विभिन्न अनुक्रमों के ट्रिपलक्स को स्थिर करने के लिए अलग-अलग आयनों की आवश्यकता होती है। चूँकि Py-Pu-Pu ट्रिपलेक्स के निर्माण के लिए उनके घटक न्यूक्लियोटाइड्स के प्रोटोनेशन की आवश्यकता नहीं होती है, ऐसे ट्रिपलक्स तटस्थ pH पर मौजूद हो सकते हैं।
ध्यान दें: प्रत्यक्ष और रिवर्स हुगस्टीन इंटरैक्शन को 1-मिथाइलथाइमिन की समरूपता द्वारा समझाया गया है: 180° के घूर्णन के परिणामस्वरूप O2 परमाणु O4 परमाणु की जगह ले लेता है, जबकि हाइड्रोजन बांड की प्रणाली संरक्षित रहती है।
दो प्रकार के ट्रिपल हेलिकॉप्टर ज्ञात हैं:
- समानांतर ट्रिपल हेलिकॉप्टर जिसमें तीसरे स्ट्रैंड की ध्रुवीयता वॉटसन-क्रिक डुप्लेक्स की होमोप्यूरिन श्रृंखला की ध्रुवीयता के साथ मेल खाती है
- एंटीपैरेलल ट्रिपल हेलिकॉप्टर, जिसमें तीसरी और होमोप्यूरिन श्रृंखला की ध्रुवताएं विपरीत होती हैं।

जी-क्वाड्रुप्लेक्स- 4-स्ट्रैंड डीएनए। यह संरचना तब बनती है जब चार गुआनिन होते हैं, जो तथाकथित जी-क्वाड्रुप्लेक्स बनाते हैं - चार गुआनिन का एक गोल नृत्य।
ऐसी संरचनाओं के निर्माण की संभावना के पहले संकेत वॉटसन और क्रिक के सफल कार्य से बहुत पहले 1910 में प्राप्त हुए थे। तब जर्मन रसायनज्ञ इवर बैंग ने पता लगाया कि डीएनए के घटकों में से एक - गुआनोसिनिक एसिड - उच्च सांद्रता पर जैल बनाता है, जबकि डीएनए के अन्य घटकों में यह गुण नहीं होता है।
1962 में, एक्स-रे विवर्तन विधि का उपयोग करके इस जेल की कोशिका संरचना स्थापित करना संभव हो गया। यह चार गुआनिन अवशेषों से बना है, जो एक दूसरे को एक वृत्त में जोड़ते हैं और एक विशिष्ट वर्ग बनाते हैं। केंद्र में, बंधन एक धातु आयन (Na, K, Mg) द्वारा समर्थित है। यदि डीएनए में बहुत अधिक ग्वानिन हो तो वही संरचनाएं डीएनए में भी बन सकती हैं। ये सपाट वर्ग (जी-चौकड़ी) काफी स्थिर, घनी संरचनाएं (जी-क्वाड्रुप्लेक्स) बनाने के लिए ढेर किए जाते हैं।
डीएनए के चार अलग-अलग स्ट्रैंड्स को चार-स्ट्रैंडेड कॉम्प्लेक्स में बुना जा सकता है, लेकिन यह एक अपवाद है। अधिक बार, न्यूक्लिक एसिड का एक एकल स्ट्रैंड बस एक गाँठ में बंध जाता है, जिससे विशिष्ट गाढ़ापन बनता है (उदाहरण के लिए, गुणसूत्रों के सिरों पर), या कुछ गुआनिन-समृद्ध क्षेत्र में डबल-स्ट्रैंडेड डीएनए एक स्थानीय चतुर्भुज बनाता है।
क्रोमोसोम के सिरों पर - टेलोमेरेस पर और ट्यूमर प्रमोटरों में - क्वाड्रप्लेक्स के अस्तित्व का सबसे अधिक अध्ययन किया गया है। हालाँकि, मानव गुणसूत्रों में ऐसे डीएनए के स्थानीयकरण की पूरी तस्वीर अभी भी ज्ञात नहीं है।
रैखिक रूप में ये सभी असामान्य डीएनए संरचनाएं बी-फॉर्म डीएनए की तुलना में अस्थिर हैं। हालाँकि, डीएनए अक्सर टोपोलॉजिकल तनाव के एक गोलाकार रूप में मौजूद होता है जब इसमें सुपरकोलिंग कहा जाता है। इन स्थितियों के तहत, गैर-विहित डीएनए संरचनाएं आसानी से बन जाती हैं: जेड-फॉर्म, "क्रॉस" और "हेयरपिन", एच-फॉर्म, गुआनिन क्वाड्रुप्लेक्स और आई-मोटिफ।
- सुपरकोइल्ड रूप - जब पेंटोस फॉस्फेट रीढ़ की हड्डी को नुकसान पहुंचाए बिना कोशिका नाभिक से जारी किया जाता है तो नोट किया जाता है। इसमें सुपर-ट्विस्टेड बंद रिंग्स का आकार है। सुपरकोइल्ड अवस्था में, डीएनए डबल हेलिक्स कम से कम एक बार "खुद पर मुड़ जाता है", यानी इसमें कम से कम एक सुपरटर्न होता है (आकृति आठ का आकार लेता है)।
- डीएनए की शिथिल अवस्था - एक ही ब्रेक (एक स्ट्रैंड का टूटना) के साथ देखी गई। इस मामले में, सुपरकॉइल्स गायब हो जाते हैं और डीएनए एक बंद रिंग का रूप ले लेता है।
- डीएनए का रैखिक रूप तब देखा जाता है जब डबल हेलिक्स की दो किस्में टूट जाती हैं।
डीएनए की तृतीयक संरचना
डीएनए की तृतीयक संरचनाएक डबल-हेलिकल अणु के स्थान में अतिरिक्त घुमाव के परिणामस्वरूप बनता है - इसकी सुपरकोलिंग। यूकेरियोटिक कोशिकाओं में डीएनए अणु की सुपरकोलिंग, प्रोकैरियोट्स के विपरीत, प्रोटीन के साथ कॉम्प्लेक्स के रूप में होती है।
यूकेरियोट्स के लगभग सभी डीएनए नाभिक के गुणसूत्रों में पाए जाते हैं; केवल थोड़ी मात्रा माइटोकॉन्ड्रिया में और पौधों में, प्लास्टिड में निहित होती है। यूकेरियोटिक कोशिकाओं (मानव गुणसूत्रों सहित) के गुणसूत्रों का मुख्य पदार्थ क्रोमैटिन है, जिसमें डबल-स्ट्रैंडेड डीएनए, हिस्टोन और गैर-हिस्टोन प्रोटीन होते हैं।
हिस्टोन क्रोमेटिन प्रोटीन
हिस्टोन सरल प्रोटीन होते हैं जो 50% तक क्रोमैटिन बनाते हैं। सभी अध्ययन किए गए जानवरों और पौधों की कोशिकाओं में, हिस्टोन के पांच मुख्य वर्ग पाए गए: H1, H2A, H2B, H3, H4, आकार, अमीनो एसिड संरचना और चार्ज (हमेशा सकारात्मक) में भिन्न।
स्तनधारी हिस्टोन H1 में एक एकल पॉलीपेप्टाइड श्रृंखला होती है जिसमें लगभग 215 अमीनो एसिड होते हैं; अन्य हिस्टोन का आकार 100 से 135 अमीनो एसिड तक भिन्न होता है। ये सभी लगभग 2.5 एनएम के व्यास के साथ एक गोलाकार में सर्पिलीकृत और मुड़े हुए हैं, और इसमें असामान्य रूप से बड़ी मात्रा में सकारात्मक रूप से चार्ज किए गए अमीनो एसिड लाइसिन और आर्जिनिन होते हैं। हिस्टोन को एसिटिलेटेड, मिथाइलेटेड, फॉस्फोराइलेटेड, पॉली (एडीपी)-राइबोसाइलेटेड किया जा सकता है, और हिस्टोन एच2ए और एच2बी सहसंयोजक रूप से यूबिकिटिन से जुड़े होते हैं। हिस्टोन द्वारा संरचना के निर्माण और कार्यों के प्रदर्शन में ऐसे संशोधनों की भूमिका अभी तक पूरी तरह से स्पष्ट नहीं की गई है। यह माना जाता है कि यह डीएनए के साथ बातचीत करने और जीन क्रिया को विनियमित करने के लिए एक तंत्र प्रदान करने की उनकी क्षमता है।
हिस्टोन मुख्य रूप से डीएनए के नकारात्मक रूप से चार्ज किए गए फॉस्फेट समूहों और हिस्टोन के सकारात्मक रूप से चार्ज किए गए लाइसिन और आर्जिनिन अवशेषों के बीच बने आयनिक बांड (नमक पुलों) के माध्यम से डीएनए के साथ बातचीत करते हैं।
गैर-हिस्टोन क्रोमैटिन प्रोटीन
हिस्टोन के विपरीत, गैर-हिस्टोन प्रोटीन बहुत विविध हैं। डीएनए-बाध्यकारी गैर-हिस्टोन प्रोटीन के 590 विभिन्न अंशों को अलग किया गया है। उन्हें अम्लीय प्रोटीन भी कहा जाता है, क्योंकि उनकी संरचना में अम्लीय अमीनो एसिड (वे पॉलीअनियन होते हैं) का प्रभुत्व होता है। गैर-हिस्टोन प्रोटीन की विविधता क्रोमैटिन गतिविधि के विशिष्ट विनियमन से जुड़ी है। उदाहरण के लिए, डीएनए प्रतिकृति और अभिव्यक्ति के लिए आवश्यक एंजाइम क्षणिक रूप से क्रोमैटिन से बंध सकते हैं। अन्य प्रोटीन, कहते हैं, जो विभिन्न नियामक प्रक्रियाओं में शामिल होते हैं, केवल विशिष्ट ऊतकों में या विभेदन के कुछ चरणों में डीएनए से जुड़ते हैं। प्रत्येक प्रोटीन डीएनए न्यूक्लियोटाइड्स (डीएनए साइट) के एक विशिष्ट अनुक्रम का पूरक है। इस समूह में शामिल हैं:
- साइट-विशिष्ट जिंक फिंगर प्रोटीन का परिवार। प्रत्येक "जिंक फिंगर" 5 न्यूक्लियोटाइड जोड़े वाली एक विशिष्ट साइट को पहचानती है।
- साइट-विशिष्ट प्रोटीन का परिवार - होमोडीमर। डीएनए के संपर्क में आने वाले ऐसे प्रोटीन के टुकड़े में हेलिक्स-टर्न-हेलिक्स संरचना होती है।
- उच्च गतिशीलता जेल प्रोटीन (एचएमजी प्रोटीन) संरचनात्मक और नियामक प्रोटीन का एक समूह है जो लगातार क्रोमैटिन से जुड़ा रहता है। इनका आणविक भार 30 kDa से कम होता है और इनमें आवेशित अमीनो एसिड की उच्च सामग्री होती है। अपने कम आणविक भार के कारण, एचएमजी प्रोटीन में पॉलीएक्रिलामाइड जेल वैद्युतकणसंचलन के दौरान उच्च गतिशीलता होती है।
- प्रतिकृति, प्रतिलेखन और मरम्मत एंजाइम।
डीएनए और आरएनए के संश्लेषण में शामिल संरचनात्मक, नियामक प्रोटीन और एंजाइमों की भागीदारी के साथ, न्यूक्लियोसोम धागा प्रोटीन और डीएनए के अत्यधिक संघनित परिसर में परिवर्तित हो जाता है। परिणामी संरचना मूल डीएनए अणु से 10,000 गुना छोटी है।

क्रोमेटिन
क्रोमैटिन परमाणु डीएनए और अकार्बनिक पदार्थों वाले प्रोटीन का एक जटिल है। क्रोमैटिन का अधिकांश भाग निष्क्रिय है। इसमें कसकर पैक किया हुआ, संघनित डीएनए होता है। यह हेटरोक्रोमैटिन है। संवैधानिक, आनुवंशिक रूप से निष्क्रिय क्रोमैटिन (उपग्रह डीएनए) होते हैं जिनमें गैर-व्यक्त क्षेत्र होते हैं, और वैकल्पिक - कई पीढ़ियों में निष्क्रिय होते हैं, लेकिन कुछ परिस्थितियों में अभिव्यक्ति में सक्षम होते हैं।
सक्रिय क्रोमैटिन (यूक्रोमैटिन) असंघनित होता है, अर्थात। कम कसकर पैक किया गया। विभिन्न कोशिकाओं में इसकी सामग्री 2 से 11% तक होती है। मस्तिष्क कोशिकाओं में यह सर्वाधिक प्रचुर मात्रा में होता है - 10-11%, यकृत कोशिकाओं में - 3-4 और गुर्दे की कोशिकाओं में - 2-3%। यूक्रोमैटिन का सक्रिय प्रतिलेखन नोट किया गया है। इसके अलावा, इसका संरचनात्मक संगठन किसी दिए गए प्रकार के जीव में निहित समान आनुवंशिक डीएनए जानकारी को विशेष कोशिकाओं में अलग-अलग तरीके से उपयोग करने की अनुमति देता है।
एक इलेक्ट्रॉन माइक्रोस्कोप में, क्रोमेटिन की छवि मोतियों से मिलती जुलती है: लगभग 10 एनएम आकार की गोलाकार मोटाई, धागे जैसे पुलों द्वारा अलग की गई। इन गोलाकार गाढ़ेपन को न्यूक्लियोसोम कहा जाता है। न्यूक्लियोसोम क्रोमैटिन की एक संरचनात्मक इकाई है। प्रत्येक न्यूक्लियोसोम में 146-बीपी सुपरकोइल्ड डीएनए खंड घाव होता है जो प्रति न्यूक्लियोसोमल कोर में 1.75 बाएं मोड़ बनाता है। न्यूक्लियोसोमल कोर एक हिस्टोन ऑक्टेमर है जिसमें हिस्टोन H2A, H2B, H3 और H4, प्रत्येक प्रकार के दो अणु होते हैं (चित्र 9), जो 11 एनएम के व्यास और 5.7 एनएम की मोटाई के साथ एक डिस्क की तरह दिखता है। पांचवां हिस्टोन, एच1, न्यूक्लियोसोमल कोर का हिस्सा नहीं है और हिस्टोन ऑक्टेमर पर डीएनए को घुमाने की प्रक्रिया में शामिल नहीं है। यह उन स्थानों पर डीएनए से संपर्क करता है जहां डबल हेलिक्स न्यूक्लियोसोमल कोर में प्रवेश करता है और बाहर निकलता है। ये इंटरकोर (लिंकर) डीएनए अनुभाग हैं, जिनकी लंबाई कोशिका प्रकार के आधार पर 40 से 50 न्यूक्लियोटाइड जोड़े तक भिन्न होती है। परिणामस्वरूप, न्यूक्लियोसोम में शामिल डीएनए टुकड़े की लंबाई भी भिन्न होती है (186 से 196 न्यूक्लियोटाइड जोड़े तक)।
न्यूक्लियोसोम में लगभग 90% डीएनए होता है, बाकी लिंकर होते हैं। ऐसा माना जाता है कि न्यूक्लियोसोम "मूक" क्रोमैटिन के टुकड़े हैं, और लिंकर सक्रिय है। हालाँकि, न्यूक्लियोसोम खुल सकते हैं और रैखिक बन सकते हैं। खुले हुए न्यूक्लियोसोम पहले से ही सक्रिय क्रोमैटिन हैं। यह संरचना पर कार्य की निर्भरता को स्पष्ट रूप से प्रदर्शित करता है। यह माना जा सकता है कि गोलाकार न्यूक्लियोसोम में जितना अधिक क्रोमैटिन होता है, वह उतना ही कम सक्रिय होता है। जाहिर है, विभिन्न कोशिकाओं में निष्क्रिय क्रोमैटिन का असमान अनुपात ऐसे न्यूक्लियोसोम की संख्या से जुड़ा होता है।
इलेक्ट्रॉन सूक्ष्म तस्वीरों में, अलगाव की स्थितियों और खिंचाव की डिग्री के आधार पर, क्रोमेटिन न केवल मोटाई के साथ एक लंबे धागे के रूप में दिख सकता है - न्यूक्लियोसोम के "मोती", बल्कि व्यास के साथ एक छोटे और सघन तंतु (फाइबर) के रूप में भी। 30 एनएम, जिसका गठन डीएनए और हिस्टोन एच3 के लिंकर क्षेत्र से बंधे हिस्टोन एच1 के इंटरेक्शन के दौरान देखा जाता है, जिससे 30 एनएम के व्यास के साथ एक सोलनॉइड बनाने के लिए प्रति मोड़ छह न्यूक्लियोसोम के हेलिक्स का अतिरिक्त घुमाव होता है। इस मामले में, हिस्टोन प्रोटीन कई जीनों के प्रतिलेखन में हस्तक्षेप कर सकता है और इस प्रकार उनकी गतिविधि को नियंत्रित कर सकता है।
ऊपर वर्णित हिस्टोन के साथ डीएनए की अंतःक्रिया के परिणामस्वरूप, 2 एनएम के औसत व्यास और 57 एनएम की लंबाई के साथ 186 बेस जोड़े के डीएनए डबल हेलिक्स का एक खंड 10 एनएम के व्यास और एक हेलिक्स में परिवर्तित हो जाता है। 5 एनएम की लंबाई. जब इस हेलिक्स को बाद में 30 एनएम के व्यास वाले फाइबर में संपीड़ित किया जाता है, तो संक्षेपण की डिग्री छह गुना बढ़ जाती है।
अंततः, पांच हिस्टोन वाले डीएनए डुप्लेक्स की पैकेजिंग के परिणामस्वरूप डीएनए का 50 गुना संघनन होता है। हालाँकि, संघनन की इतनी उच्च डिग्री भी मेटाफ़ेज़ गुणसूत्र में डीएनए के लगभग 50,000 - 100,000 गुना संघनन की व्याख्या नहीं कर सकती है। दुर्भाग्य से, मेटाफ़ेज़ क्रोमोसोम तक आगे क्रोमैटिन पैकेजिंग का विवरण अभी तक ज्ञात नहीं है, इसलिए हम केवल इस प्रक्रिया की सामान्य विशेषताओं पर विचार कर सकते हैं।
गुणसूत्रों में डीएनए संघनन का स्तर
प्रत्येक डीएनए अणु को एक अलग गुणसूत्र में पैक किया जाता है। मानव द्विगुणित कोशिकाओं में 46 गुणसूत्र होते हैं, जो कोशिका केन्द्रक में स्थित होते हैं। एक कोशिका में सभी गुणसूत्रों के डीएनए की कुल लंबाई 1.74 मीटर है, लेकिन नाभिक का व्यास जिसमें गुणसूत्र पैक होते हैं, लाखों गुना छोटा होता है। कोशिका नाभिक में गुणसूत्रों और गुणसूत्रों में डीएनए की ऐसी कॉम्पैक्ट पैकेजिंग विभिन्न प्रकार के हिस्टोन और गैर-हिस्टोन प्रोटीन द्वारा सुनिश्चित की जाती है जो डीएनए के साथ एक निश्चित अनुक्रम में बातचीत करते हैं (ऊपर देखें)। गुणसूत्रों में डीएनए को संकुचित करने से इसके रैखिक आयामों को लगभग 10,000 गुना - लगभग 5 सेमी से 5 माइक्रोन तक कम करना संभव हो जाता है। संघनन के कई स्तर हैं (चित्र 10)।


- डीएनए डबल हेलिक्स एक नकारात्मक चार्ज अणु है जिसका व्यास 2 एनएम और लंबाई कई सेमी है।
- न्यूक्लियोसोम स्तर- क्रोमैटिन एक इलेक्ट्रॉन माइक्रोस्कोप में "मोतियों" की एक श्रृंखला के रूप में दिखता है - न्यूक्लियोसोम - "एक धागे पर"। न्यूक्लियोसोम एक सार्वभौमिक संरचनात्मक इकाई है जो इंटरफेज़ न्यूक्लियस और मेटाफ़ेज़ क्रोमोसोम में यूक्रोमैटिन और हेटरोक्रोमैटिन दोनों में पाई जाती है।
संघनन का न्यूक्लियोसोमल स्तर विशेष प्रोटीन - हिस्टोन द्वारा सुनिश्चित किया जाता है। आठ सकारात्मक रूप से चार्ज किए गए हिस्टोन डोमेन न्यूक्लियोसोम के मूल का निर्माण करते हैं जिसके चारों ओर एक नकारात्मक चार्ज वाला डीएनए अणु घाव होता है। इससे 7 गुना छोटापन मिलता है, जबकि व्यास 2 से 11 एनएम तक बढ़ जाता है।
- सोलनॉइड स्तर
गुणसूत्र संगठन के सोलनॉइड स्तर को न्यूक्लियोसोम फिलामेंट के मुड़ने और 20-35 एनएम व्यास वाले मोटे तंतुओं के गठन की विशेषता है - सोलनॉइड या सुपरबिड। सोलनॉइड पिच 11 एनएम है; प्रति मोड़ लगभग 6-10 न्यूक्लियोसोम होते हैं। सोलेनॉइड पैकिंग को सुपरबिड पैकिंग की तुलना में अधिक संभावित माना जाता है, जिसके अनुसार 20-35 एनएम के व्यास वाला एक क्रोमैटिन फाइब्रिल ग्रैन्यूल या सुपरबिड की एक श्रृंखला है, जिनमें से प्रत्येक में आठ न्यूक्लियोसोम होते हैं। सोलनॉइड स्तर पर, डीएनए का रैखिक आकार 6-10 गुना कम हो जाता है, व्यास 30 एनएम तक बढ़ जाता है।
- पाश स्तर
लूप स्तर गैर-हिस्टोन साइट-विशिष्ट डीएनए-बाध्यकारी प्रोटीन द्वारा प्रदान किया जाता है जो विशिष्ट डीएनए अनुक्रमों को पहचानते हैं और बांधते हैं, जिससे लगभग 30-300 केबी के लूप बनते हैं। लूप जीन अभिव्यक्ति सुनिश्चित करता है, अर्थात लूप न केवल एक संरचनात्मक, बल्कि एक कार्यात्मक गठन भी है। इस स्तर पर लघुकरण 20-30 बार होता है। व्यास 300 एनएम तक बढ़ जाता है। उभयचर ओसाइट्स में "लैंप ब्रश" जैसी लूप-आकार की संरचनाएं साइटोलॉजिकल तैयारियों में देखी जा सकती हैं। ये लूप सुपरकॉइल्ड प्रतीत होते हैं और डीएनए डोमेन का प्रतिनिधित्व करते हैं, जो संभवतः प्रतिलेखन और क्रोमैटिन प्रतिकृति की इकाइयों के अनुरूप होते हैं। विशिष्ट प्रोटीन लूप के आधारों और, संभवतः, उनके कुछ आंतरिक वर्गों को ठीक करते हैं। लूप जैसा डोमेन संगठन मेटाफ़ेज़ गुणसूत्रों में क्रोमैटिन को उच्च क्रम की पेचदार संरचनाओं में मोड़ने को बढ़ावा देता है।
- डोमेन स्तर
गुणसूत्र संगठन के डोमेन स्तर का पर्याप्त अध्ययन नहीं किया गया है। इस स्तर पर, लूप डोमेन का गठन नोट किया जाता है - 25-30 एनएम मोटे धागे (फाइब्रिल्स) की संरचनाएं, जिनमें 60% प्रोटीन, 35% डीएनए और 5% आरएनए होते हैं, कोशिका चक्र के सभी चरणों में व्यावहारिक रूप से अदृश्य होते हैं। माइटोसिस का अपवाद है और कुछ हद तक कोशिका केन्द्रक में अनियमित रूप से वितरित होता है। उभयचर ओसाइट्स में "लैंप ब्रश" जैसी लूप-आकार की संरचनाएं साइटोलॉजिकल तैयारियों में देखी जा सकती हैं।
लूप डोमेन अपने आधार पर तथाकथित अंतर्निहित अनुलग्नक साइटों में इंट्रान्यूक्लियर प्रोटीन मैट्रिक्स से जुड़े होते हैं, जिन्हें अक्सर MAR/SAR अनुक्रम (MAR, अंग्रेजी मैट्रिक्स संबद्ध क्षेत्र से; SAR, अंग्रेजी मचान अनुलग्नक क्षेत्रों से) कहा जाता है। - डीएनए कई सौ लंबाई वाले बेस जोड़े के टुकड़े करता है, जिनमें ए/टी न्यूक्लियोटाइड जोड़े की उच्च सामग्री (>65%) होती है। ऐसा प्रतीत होता है कि प्रत्येक डोमेन में प्रतिकृति की एक ही उत्पत्ति होती है और यह एक स्वायत्त सुपरहेलिकल इकाई के रूप में कार्य करता है। किसी भी लूप डोमेन में कई प्रतिलेखन इकाइयाँ होती हैं, जिनकी कार्यप्रणाली संभवतः समन्वित होती है - संपूर्ण डोमेन या तो सक्रिय या निष्क्रिय स्थिति में होता है।
डोमेन स्तर पर, अनुक्रमिक क्रोमैटिन पैकेजिंग के परिणामस्वरूप, डीएनए के रैखिक आयामों में लगभग 200 गुना (700 एनएम) की कमी होती है।
- गुणसूत्र स्तर
क्रोमोसोमल स्तर पर, प्रोफ़ेज़ क्रोमोसोम का मेटाफ़ेज़ क्रोमोसोम में संघनन गैर-हिस्टोन प्रोटीन के अक्षीय ढांचे के आसपास लूप डोमेन के संघनन के साथ होता है। यह सुपरकोलिंग कोशिका में सभी H1 अणुओं के फॉस्फोराइलेशन के साथ होता है। परिणामस्वरूप, मेटाफ़ेज़ गुणसूत्र को घने सर्पिल में कुंडलित, घनीभूत सोलनॉइड लूप के रूप में चित्रित किया जा सकता है। एक सामान्य मानव गुणसूत्र में 2,600 लूप तक हो सकते हैं। ऐसी संरचना की मोटाई 1400 एनएम (दो क्रोमैटिड) तक पहुंच जाती है, और डीएनए अणु 104 गुना छोटा हो जाता है, यानी। 5 सेमी से 5 µm तक फैला हुआ डीएनए।
गुणसूत्रों के कार्य
एक्स्ट्राक्रोमोसोमल तंत्र के साथ बातचीत में, गुणसूत्र प्रदान करते हैं
- वंशानुगत जानकारी का भंडारण
- सेलुलर संगठन बनाने और बनाए रखने के लिए इस जानकारी का उपयोग करना
- वंशानुगत जानकारी पढ़ने का विनियमन
- आनुवंशिक सामग्री का स्व-दोहराव
- मातृ कोशिका से पुत्री कोशिकाओं में आनुवंशिक सामग्री का स्थानांतरण।
इस बात के प्रमाण हैं कि जब क्रोमैटिन का एक क्षेत्र सक्रिय होता है, अर्थात। प्रतिलेखन के दौरान, पहले हिस्टोन एच1 और फिर हिस्टोन ऑक्टेट को इससे उलटा हटा दिया जाता है। यह क्रोमैटिन डिकॉन्डेंसेशन का कारण बनता है, 30-एनएम क्रोमैटिन फाइब्रिल का 10-एनएम फाइब्रिल में अनुक्रमिक संक्रमण और इसके आगे मुक्त डीएनए के वर्गों में खुलासा होता है, यानी। न्यूक्लियोसोम संरचना का नुकसान।
मॉस्को, 25 अप्रैल - आरआईए नोवोस्ती, तात्याना पिचुगिना।ठीक 65 साल पहले, ब्रिटिश वैज्ञानिकों जेम्स वॉटसन और फ्रांसिस क्रिक ने डीएनए की संरचना को समझने, एक नए विज्ञान - आणविक जीव विज्ञान की नींव रखने पर एक लेख प्रकाशित किया था। इस खोज ने मानव जाति के जीवन में बहुत कुछ बदल दिया। आरआईए नोवोस्ती डीएनए अणु के गुणों के बारे में बात करती है और यह इतना महत्वपूर्ण क्यों है।
19वीं शताब्दी के उत्तरार्ध में, जीव विज्ञान एक बहुत ही युवा विज्ञान था। वैज्ञानिक अभी कोशिका का अध्ययन करना शुरू ही कर रहे थे, और आनुवंशिकता के बारे में विचार, हालांकि पहले से ही ग्रेगर मेंडल द्वारा तैयार किए गए थे, व्यापक रूप से स्वीकार नहीं किए गए थे।
1868 के वसंत में, एक युवा स्विस डॉक्टर, फ्रेडरिक मिशर, वैज्ञानिक कार्य में संलग्न होने के लिए तुबिंगन विश्वविद्यालय (जर्मनी) पहुंचे। उनका इरादा यह पता लगाना था कि कोशिका किन पदार्थों से बनी होती है। प्रयोगों के लिए मैंने ल्यूकोसाइट्स को चुना, जो मवाद से प्राप्त करना आसान है।
नाभिक को प्रोटोप्लाज्म, प्रोटीन और वसा से अलग करते हुए, मिशर ने उच्च फास्फोरस सामग्री वाले एक यौगिक की खोज की। उन्होंने इस अणु को न्यूक्लिन (लैटिन में "नाभिक") - न्यूक्लियस) कहा।
इस यौगिक ने अम्लीय गुण प्रदर्शित किए, यही कारण है कि "न्यूक्लिक एसिड" शब्द उत्पन्न हुआ। इसके उपसर्ग "डीऑक्सीराइबो" का अर्थ है कि अणु में एच-समूह और शर्करा हैं। तब पता चला कि यह वास्तव में नमक था, लेकिन उन्होंने नाम नहीं बदला।
20वीं सदी की शुरुआत में, वैज्ञानिकों को पहले से ही पता था कि न्यूक्लिन एक बहुलक है (अर्थात, दोहराई जाने वाली इकाइयों का एक बहुत लंबा लचीला अणु), इकाइयाँ चार नाइट्रोजनस आधारों (एडेनिन, थाइमिन, गुआनिन और साइटोसिन) और न्यूक्लिन से बनी होती हैं। गुणसूत्रों में निहित है - कॉम्पैक्ट संरचनाएं जो विभाजित कोशिकाओं में होती हैं। वंशानुगत विशेषताओं को प्रसारित करने की उनकी क्षमता का प्रदर्शन अमेरिकी आनुवंशिकीविद् थॉमस मॉर्गन ने फल मक्खियों पर प्रयोगों में किया था।
वह मॉडल जिसने जीन की व्याख्या की
लेकिन डीऑक्सीराइबोन्यूक्लिक एसिड, या संक्षेप में डीएनए, कोशिका नाभिक में क्या करता है, यह लंबे समय से समझ में नहीं आया है। ऐसा माना जाता था कि यह गुणसूत्रों में कुछ संरचनात्मक भूमिका निभाता है। आनुवंशिकता की इकाइयों - जीन - को प्रोटीन प्रकृति के लिए जिम्मेदार ठहराया गया था। यह सफलता अमेरिकी शोधकर्ता ओसवाल्ड एवरी द्वारा की गई, जिन्होंने प्रयोगात्मक रूप से साबित किया कि आनुवंशिक सामग्री डीएनए के माध्यम से बैक्टीरिया से बैक्टीरिया में स्थानांतरित होती है।
यह स्पष्ट हो गया कि डीएनए का अध्ययन करने की आवश्यकता है। आख़िर कैसे? उस समय वैज्ञानिकों के लिए केवल एक्स-रे ही उपलब्ध थे। इसके साथ जैविक अणुओं को रोशन करने के लिए, उन्हें क्रिस्टलीकृत करना होगा, और यह मुश्किल है। कैवेंडिश प्रयोगशाला (कैम्ब्रिज, यूके) में एक्स-रे विवर्तन पैटर्न से प्रोटीन अणुओं की संरचना को समझा गया था। वहां काम करने वाले युवा शोधकर्ताओं, जेम्स वॉटसन और फ्रांसिस क्रिक के पास डीएनए पर अपना स्वयं का प्रयोगात्मक डेटा नहीं था, इसलिए उन्होंने किंग्स कॉलेज मौरिस विल्किंस और रोज़लिंड फ्रैंकलिन के सहयोगियों की एक्स-रे तस्वीरों का इस्तेमाल किया।
वॉटसन और क्रिक ने डीएनए संरचना का एक मॉडल प्रस्तावित किया जो एक्स-रे पैटर्न से बिल्कुल मेल खाता था: दो समानांतर किस्में दाएं हाथ के हेलिक्स में मुड़ गईं। प्रत्येक श्रृंखला उनके शर्करा और फॉस्फेट की रीढ़ पर फंसे नाइट्रोजनस आधारों के एक यादृच्छिक सेट से बनी होती है, और आधारों के बीच हाइड्रोजन बांड द्वारा एक साथ रखी जाती है। इसके अलावा, एडेनिन केवल थाइमिन के साथ और गुआनिन साइटोसिन के साथ जुड़ता है। इस नियम को संपूरकता का सिद्धांत कहा जाता है।
वॉटसन और क्रिक मॉडल ने डीएनए के चार मुख्य कार्यों की व्याख्या की: आनुवंशिक सामग्री की प्रतिकृति, इसकी विशिष्टता, अणु में जानकारी का भंडारण, और इसकी उत्परिवर्तन करने की क्षमता।
वैज्ञानिकों ने अपनी खोज को 25 अप्रैल, 1953 को नेचर पत्रिका में प्रकाशित किया। दस साल बाद, मौरिस विल्किंस के साथ, उन्हें जीव विज्ञान में नोबेल पुरस्कार से सम्मानित किया गया (रोज़ालिंड फ्रैंकलिन की 1958 में 37 वर्ष की आयु में कैंसर से मृत्यु हो गई)।
"अब, आधी सदी से भी अधिक समय के बाद, हम कह सकते हैं कि डीएनए की संरचना की खोज ने जीव विज्ञान के विकास में वही भूमिका निभाई जो भौतिकी में परमाणु नाभिक की खोज ने निभाई। परमाणु की संरचना की व्याख्या ने एक उत्कृष्ट आनुवंशिकीविद्, डीएनए शोधकर्ता और पुस्तक "द सबसे महत्वपूर्ण अणु।”
जेनेटिक कोड
अब बस यह पता लगाना बाकी रह गया था कि यह अणु कैसे काम करता है। यह ज्ञात था कि डीएनए में सेलुलर प्रोटीन के संश्लेषण के लिए निर्देश होते हैं, जो कोशिका में सभी कार्य करते हैं। प्रोटीन अमीनो एसिड के दोहराए जाने वाले सेट (अनुक्रम) से बने पॉलिमर हैं। इसके अलावा, केवल बीस अमीनो एसिड होते हैं। जानवरों की प्रजातियाँ अपनी कोशिकाओं में प्रोटीन के सेट में, यानी अलग-अलग अमीनो एसिड अनुक्रम में एक-दूसरे से भिन्न होती हैं। आनुवंशिकीविदों ने दावा किया कि ये अनुक्रम जीन द्वारा निर्धारित किए गए थे, जिन्हें तब जीवन के निर्माण खंड के रूप में माना जाता था। लेकिन कोई नहीं जानता था कि वास्तव में जीन क्या होते हैं।
स्पष्टता बिग बैंग सिद्धांत के लेखक, जॉर्ज वाशिंगटन विश्वविद्यालय (यूएसए) के एक कर्मचारी, भौतिक विज्ञानी जॉर्जी गामो द्वारा लाई गई थी। वॉटसन और क्रिक के डबल-स्ट्रैंडेड डीएनए हेलिक्स के मॉडल के आधार पर, उन्होंने सुझाव दिया कि एक जीन डीएनए का एक खंड है, यानी लिंक का एक निश्चित अनुक्रम - न्यूक्लियोटाइड। चूँकि प्रत्येक न्यूक्लियोटाइड चार नाइट्रोजनस आधारों में से एक है, हमें बस यह पता लगाने की आवश्यकता है कि चार तत्व बीस के लिए कैसे कोड करते हैं। यह जेनेटिक कोड का विचार था.
1960 के दशक की शुरुआत में, यह स्थापित हो गया था कि प्रोटीन को राइबोसोम में अमीनो एसिड से संश्लेषित किया जाता है, जो कोशिका के अंदर एक प्रकार का "कारखाना" है। प्रोटीन संश्लेषण शुरू करने के लिए, एक एंजाइम डीएनए के पास जाता है, जीन की शुरुआत में एक निश्चित क्षेत्र को पहचानता है, जीन की एक प्रति को छोटे आरएनए (इसे टेम्पलेट कहा जाता है) के रूप में संश्लेषित करता है, फिर प्रोटीन राइबोसोम में विकसित होता है अमीनो अम्ल।
उन्होंने यह भी पता लगाया कि आनुवंशिक कोड तीन अक्षरों का होता है। इसका मतलब है कि एक अमीनो एसिड तीन न्यूक्लियोटाइड से मेल खाता है। कोड की इकाई को कोडन कहा जाता है। राइबोसोम में, एमआरएनए से जानकारी क्रमिक रूप से कोडन द्वारा पढ़ी जाती है। और उनमें से प्रत्येक कई अमीनो एसिड से मेल खाता है। सिफर कैसा दिखता है?
इस प्रश्न का उत्तर संयुक्त राज्य अमेरिका के मार्शल निरेनबर्ग और हेनरिक मैटेई ने दिया। 1961 में, उन्होंने पहली बार मास्को में जैव रासायनिक कांग्रेस में अपने परिणामों की सूचना दी। 1967 तक, आनुवंशिक कोड को पूरी तरह से समझ लिया गया था। यह सभी जीवों की सभी कोशिकाओं के लिए सार्वभौमिक साबित हुआ, जिसके विज्ञान के लिए दूरगामी परिणाम हुए।
डीएनए की संरचना और आनुवंशिक कोड की खोज ने जैविक अनुसंधान को पूरी तरह से पुनर्निर्देशित कर दिया। तथ्य यह है कि प्रत्येक व्यक्ति के पास एक अद्वितीय डीएनए अनुक्रम होता है जिसने फोरेंसिक विज्ञान में क्रांति ला दी है। मानव जीनोम को समझने से मानवविज्ञानियों को हमारी प्रजातियों के विकास का अध्ययन करने के लिए एक पूरी तरह से नई विधि मिल गई है। हाल ही में आविष्कार किए गए डीएनए संपादक सीआरआईएसपीआर-कैस ने जेनेटिक इंजीनियरिंग को काफी उन्नत किया है। जाहिर है, इस अणु में मानवता की सबसे गंभीर समस्याओं का समाधान शामिल है: कैंसर, आनुवंशिक रोग, उम्र बढ़ना।
डीएनए की संरचना और कार्य
| मापदण्ड नाम | अर्थ |
| लेख का विषय: | डीएनए की संरचना और कार्य |
| रूब्रिक (विषयगत श्रेणी) | शिक्षा |
डीएनए- एक बहुलक जिसके मोनोमर्स डीऑक्सीराइबोन्यूक्लियोटाइड्स होते हैं। डबल हेलिक्स के रूप में डीएनए अणु की स्थानिक संरचना का एक मॉडल 1953 में प्रस्तावित किया गया था। जे. वाटसन और एफ. क्रिक (इस मॉडल को बनाने के लिए उन्होंने एम. विल्किंस, आर. फ्रैंकलिन, ई. चारगफ के कार्यों का उपयोग किया)।
डीएनए अणुदो पॉलीन्यूक्लियोटाइड श्रृंखलाओं द्वारा निर्मित, एक दूसरे के चारों ओर और एक साथ एक काल्पनिक अक्ष के चारों ओर सर्पिल रूप से मुड़े हुए, ᴛ.ᴇ। एक डबल हेलिक्स है (कुछ डीएनए युक्त वायरस के अपवाद के साथ एकल-फंसे डीएनए होते हैं)। डीएनए डबल हेलिक्स का व्यास 2 एनएम है, पड़ोसी न्यूक्लियोटाइड्स के बीच की दूरी 0.34 एनएम है, और हेलिक्स के प्रति मोड़ में 10 न्यूक्लियोटाइड जोड़े हैं। अणु की लंबाई कई सेंटीमीटर तक पहुंच सकती है। आणविक भार - दसियों और करोड़ों। मानव कोशिका के केंद्रक में डीएनए की कुल लंबाई लगभग 2 मीटर होती है। यूकेरियोटिक कोशिकाओं में, डीएनए प्रोटीन के साथ कॉम्प्लेक्स बनाता है और इसमें एक विशिष्ट स्थानिक संरचना होती है।
डीएनए मोनोमर - न्यूक्लियोटाइड (डीऑक्सीराइबोन्यूक्लियोटाइड)- इसमें तीन पदार्थों के अवशेष होते हैं: 1) एक नाइट्रोजनस बेस, 2) एक पांच-कार्बन मोनोसेकेराइड (पेंटोस) और 3) फॉस्फोरिक एसिड। न्यूक्लिक एसिड के नाइट्रोजनस आधार पाइरीमिडीन और प्यूरीन के वर्गों से संबंधित हैं। डीएनए पिरिमिडीन आधार(उनके अणु में एक वलय होता है) - थाइमिन, साइटोसिन। प्यूरीन आधार(दो वलय हैं) - एडेनिन और गुआनिन।
डीएनए न्यूक्लियोटाइड मोनोसैकेराइड डीऑक्सीराइबोज़ है।
न्यूक्लियोटाइड का नाम संबंधित आधार के नाम से लिया गया है। न्यूक्लियोटाइड और नाइट्रोजनस आधारों को बड़े अक्षरों में दर्शाया जाता है।
पॉलीन्यूक्लियोटाइड श्रृंखला न्यूक्लियोटाइड संघनन प्रतिक्रियाओं के परिणामस्वरूप बनती है। इस मामले में, एक न्यूक्लियोटाइड के डीऑक्सीराइबोज अवशेष के 3"-कार्बन और दूसरे के फॉस्फोरिक एसिड अवशेष के बीच, फॉस्फोएस्टर बंधन(मजबूत सहसंयोजक बंधों की श्रेणी के अंतर्गत आता है)। पॉलीन्यूक्लियोटाइड श्रृंखला का एक सिरा 5" कार्बन (जिसे 5" सिरा कहा जाता है) के साथ समाप्त होता है, दूसरा सिरा 3" कार्बन (3" सिरा) के साथ समाप्त होता है।
न्यूक्लियोटाइड्स के एक स्ट्रैंड के विपरीत दूसरा स्ट्रैंड होता है। इन दो श्रृंखलाओं में न्यूक्लियोटाइड की व्यवस्था यादृच्छिक नहीं है, लेकिन कड़ाई से परिभाषित है: थाइमिन हमेशा दूसरी श्रृंखला में एक श्रृंखला के एडेनिन के विपरीत स्थित होता है, और साइटोसिन हमेशा ग्वानिन के विपरीत स्थित होता है, एडेनिन और थाइमिन के बीच और बीच में दो हाइड्रोजन बंधन उत्पन्न होते हैं गुआनिन और साइटोसिन - तीन हाइड्रोजन बांड। वह पैटर्न जिसके अनुसार विभिन्न डीएनए श्रृंखलाओं के न्यूक्लियोटाइड सख्ती से व्यवस्थित होते हैं (एडेनिन - थाइमिन, गुआनिन - साइटोसिन) और चयनात्मक रूप से एक दूसरे से जुड़ते हैं, आमतौर पर कहा जाता है संपूरकता का सिद्धांत. यह ध्यान दिया जाना चाहिए कि जे. वाटसन और एफ. क्रिक को ई. चारगफ के कार्यों से परिचित होने के बाद पूरकता के सिद्धांत की समझ आई। ई. चारगफ़ ने विभिन्न जीवों के ऊतकों और अंगों के बड़ी संख्या में नमूनों का अध्ययन करते हुए पाया कि किसी भी डीएनए टुकड़े में ग्वानिन अवशेषों की सामग्री हमेशा साइटोसिन की सामग्री से मेल खाती है, और एडेनिन थाइमिन से मेल खाती है ( "चारगफ़ का नियम"), लेकिन वह इस तथ्य को समझाने में असमर्थ है।
संपूरकता के सिद्धांत से यह निष्कर्ष निकलता है कि एक श्रृंखला का न्यूक्लियोटाइड अनुक्रम दूसरे के न्यूक्लियोटाइड अनुक्रम को निर्धारित करता है।
डीएनए स्ट्रैंड एंटीपैरेलल (बहुदिशात्मक), ᴛ.ᴇ हैं। विभिन्न श्रृंखलाओं के न्यूक्लियोटाइड विपरीत दिशाओं में स्थित होते हैं, और इसलिए, एक श्रृंखला के 3" सिरे के विपरीत दूसरे का 5" सिरा होता है। डीएनए अणु की तुलना कभी-कभी सर्पिल सीढ़ी से की जाती है। इस सीढ़ी की "रेलिंग" एक शुगर-फॉस्फेट बैकबोन (डीऑक्सीराइबोज और फॉस्फोरिक एसिड के वैकल्पिक अवशेष) है; "चरण" पूरक नाइट्रोजनी आधार हैं।
डीएनए का कार्य- वंशानुगत जानकारी का भंडारण और प्रसारण।
डीएनए की संरचना और कार्य - अवधारणा और प्रकार। "डीएनए की संरचना और कार्य" 2017, 2018 श्रेणी का वर्गीकरण और विशेषताएं।
डीएनए एक सार्वभौमिक स्रोत और वंशानुगत जानकारी का रक्षक है, जिसे न्यूक्लियोटाइड के एक विशेष अनुक्रम का उपयोग करके दर्ज किया जाता है; यह सभी जीवित जीवों के गुणों को निर्धारित करता है।
एक न्यूक्लियोटाइड का औसत आणविक भार 345 माना जाता है, और न्यूक्लियोटाइड अवशेषों की संख्या कई सौ, हजार और यहां तक कि लाखों तक पहुंच सकती है। डीएनए अधिकतर कोशिकाओं के केंद्रक में पाया जाता है। क्लोरोप्लास्ट और माइटोकॉन्ड्रिया में थोड़ा सा पाया जाता है। हालाँकि, कोशिका केन्द्रक का डीएनए एक अणु नहीं है। इसमें कई अणु होते हैं जो विभिन्न गुणसूत्रों पर वितरित होते हैं, उनकी संख्या जीव के आधार पर भिन्न होती है। ये डीएनए की संरचनात्मक विशेषताएं हैं।
डीएनए की खोज का इतिहास
डीएनए की संरचना और कार्यों की खोज जेम्स वॉटसन और फ्रांसिस क्रिक ने की थी और उन्हें 1962 में नोबेल पुरस्कार से भी सम्मानित किया गया था।

लेकिन जर्मनी में काम करने वाले स्विस वैज्ञानिक फ्रेडरिक जोहान मिशर न्यूक्लिक एसिड की खोज करने वाले पहले व्यक्ति थे। 1869 में, उन्होंने पशु कोशिकाओं - ल्यूकोसाइट्स का अध्ययन किया। उन्हें प्राप्त करने के लिए, उन्होंने मवाद वाली पट्टियों का उपयोग किया, जो उन्हें अस्पतालों से मिलीं। मिशर ने मवाद से ल्यूकोसाइट्स को धोया और उनसे प्रोटीन अलग किया। इन अध्ययनों के दौरान, वैज्ञानिक यह स्थापित करने में सक्षम थे कि ल्यूकोसाइट्स में, प्रोटीन के अलावा, कुछ और भी है, कुछ पदार्थ जो उस समय अज्ञात थे। यह एक धागे जैसा या फ्लोक्यूलेंट तलछट था जो अम्लीय वातावरण बनने पर निकलता था। क्षार मिलाने पर अवक्षेप तुरंत घुल गया।

माइक्रोस्कोप का उपयोग करते हुए, वैज्ञानिक ने पाया कि जब ल्यूकोसाइट्स को हाइड्रोक्लोरिक एसिड से धोया जाता है, तो कोशिकाओं से नाभिक बच जाते हैं। तब उन्होंने निष्कर्ष निकाला कि नाभिक में एक अज्ञात पदार्थ था, जिसे उन्होंने न्यूक्लिन कहा (अनुवाद में नाभिक शब्द का अर्थ नाभिक होता है)।
रासायनिक विश्लेषण करने के बाद मिशर को पता चला कि नए पदार्थ में कार्बन, हाइड्रोजन, ऑक्सीजन और फास्फोरस शामिल हैं। उस समय, ऑर्गेनोफॉस्फोरस यौगिकों के बारे में बहुत कम जानकारी थी, इसलिए फ्रेडरिक का मानना था कि उन्होंने कोशिका नाभिक में पाए जाने वाले यौगिकों के एक नए वर्ग की खोज की थी।
इस प्रकार, 19वीं शताब्दी में न्यूक्लिक एसिड के अस्तित्व की खोज की गई। हालाँकि, उस समय कोई भी उनके द्वारा निभाई गई महत्वपूर्ण भूमिका के बारे में सोच भी नहीं सकता था।
आनुवंशिकता का पदार्थ
डीएनए की संरचना का अध्ययन जारी रहा और 1944 में ओसवाल्ड एवरी के नेतृत्व में जीवाणुविज्ञानी के एक समूह को सबूत मिला कि यह अणु गंभीर ध्यान देने योग्य है। वैज्ञानिक ने कई वर्षों तक न्यूमोकोकी, जीवों का अध्ययन किया जो निमोनिया या फेफड़ों की बीमारी का कारण बनते हैं। एवरी ने रोग पैदा करने वाले न्यूमोकोकी को जीवित जीवों के लिए सुरक्षित पदार्थों के साथ मिलाकर प्रयोग किए। सबसे पहले, रोग पैदा करने वाली कोशिकाओं को मार दिया गया, और फिर जो रोग पैदा नहीं करते थे, उन्हें उनमें जोड़ दिया गया।
शोध के नतीजों ने सभी को हैरान कर दिया. वहाँ जीवित कोशिकाएँ थीं, जो मृत कोशिकाओं के साथ बातचीत करने के बाद बीमारी पैदा करना सीख गईं। वैज्ञानिक ने उस पदार्थ की प्रकृति का पता लगाया जो मृत कोशिकाओं से जीवित कोशिकाओं तक सूचना प्रसारित करने की प्रक्रिया में शामिल है। डीएनए अणु यह पदार्थ निकला।

संरचना
इसलिए, यह समझना आवश्यक है कि डीएनए अणु की संरचना क्या है। इसकी संरचना की खोज एक महत्वपूर्ण घटना थी; इससे आणविक जीव विज्ञान का निर्माण हुआ - जैव रसायन की एक नई शाखा। डीएनए कोशिकाओं के नाभिक में बड़ी मात्रा में पाया जाता है, लेकिन अणुओं का आकार और संख्या जीव के प्रकार पर निर्भर करती है। यह स्थापित किया गया है कि स्तनधारी कोशिकाओं के नाभिक में इनमें से कई कोशिकाएं होती हैं, वे गुणसूत्रों के साथ वितरित होती हैं, उनमें से 46 हैं।
डीएनए की संरचना का अध्ययन करते हुए, 1924 में फ़्यूलगेन ने पहली बार इसका स्थानीयकरण स्थापित किया। प्रयोगों से प्राप्त साक्ष्यों से पता चला कि डीएनए माइटोकॉन्ड्रिया (1-2%) में स्थित है। अन्यत्र, ये अणु वायरल संक्रमण के दौरान, बेसल शरीर में और कुछ जानवरों के अंडों में भी पाए जा सकते हैं। यह ज्ञात है कि जीव जितना अधिक जटिल होगा, डीएनए का द्रव्यमान उतना ही अधिक होगा। किसी कोशिका में मौजूद अणुओं की संख्या उसके कार्य पर निर्भर करती है और आमतौर पर 1-10% होती है। उनमें से सबसे कम मायोसाइट्स (0.2%) में पाया जाता है, सबसे अधिक रोगाणु कोशिकाओं (60%) में पाया जाता है।

डीएनए की संरचना से पता चला है कि उच्च जीवों के गुणसूत्रों में वे सरल प्रोटीन - एल्ब्यूमिन, हिस्टोन और अन्य से जुड़े होते हैं, जो मिलकर डीएनपी (डीऑक्सीराइबोन्यूक्लियोप्रोटीन) बनाते हैं। आमतौर पर, एक बड़ा अणु अस्थिर होता है, और विकास के दौरान इसके बरकरार और अपरिवर्तित रहने के लिए, एक तथाकथित मरम्मत प्रणाली बनाई गई है, जिसमें एंजाइम - लिगेज और न्यूक्लियस शामिल हैं, जो "मरम्मत" के लिए जिम्मेदार हैं। अणु.
डीएनए की रासायनिक संरचना
डीएनए एक बहुलक, एक पॉलीन्यूक्लियोटाइड है, जिसमें बड़ी संख्या में (हज़ारों लाखों तक) मोनोन्यूक्लियोटाइड होते हैं। डीएनए की संरचना इस प्रकार है: मोनोन्यूक्लियोटाइड में नाइट्रोजनस आधार होते हैं - साइटोसिन (सी) और थाइमिन (टी) - पाइरीमिडीन डेरिवेटिव से, एडेनिन (ए) और गुआनिन (जी) - प्यूरीन डेरिवेटिव से। नाइट्रोजनस आधारों के अलावा, मानव और पशु अणु में 5-मिथाइलसिटोसिन, एक छोटा पिरिमिडीन आधार होता है। नाइट्रोजनस क्षार फॉस्फोरिक एसिड और डीऑक्सीराइबोज से बंधते हैं। डीएनए की संरचना नीचे दिखाई गई है।

चारगफ़ नियम
डीएनए की संरचना और जैविक भूमिका का अध्ययन ई. चारगफ ने 1949 में किया था। अपने शोध के दौरान, उन्होंने ऐसे पैटर्न की पहचान की जो नाइट्रोजनस आधारों के मात्रात्मक वितरण में देखे गए थे:
- ∑T + C = ∑A + G (अर्थात, पाइरीमिडीन आधारों की संख्या प्यूरीन आधारों की संख्या के बराबर है)।
- एडेनिन अवशेषों की संख्या हमेशा थाइमिन अवशेषों की संख्या के बराबर होती है, और ग्वानिन की संख्या साइटोसिन के बराबर होती है।
- विशिष्टता गुणांक का सूत्र है: G+C/A+T. उदाहरण के लिए, एक व्यक्ति के लिए यह 1.5 है, एक बैल के लिए यह 1.3 है।
- "ए + सी" का योग "जी + टी" के योग के बराबर है, यानी इसमें ग्वानिन और थाइमिन जितना ही एडेनिन और साइटोसिन होता है।
डीएनए संरचना मॉडल
इसे वॉटसन और क्रिक ने बनाया था। फॉस्फेट और डीऑक्सीराइबोज अवशेष सर्पिल तरीके से मुड़ी हुई दो पॉलीन्यूक्लियोटाइड श्रृंखलाओं की रीढ़ की हड्डी के साथ स्थित होते हैं। यह निर्धारित किया गया था कि पाइरीमिडीन और प्यूरीन आधारों की समतल संरचनाएं श्रृंखला अक्ष के लंबवत स्थित होती हैं और सर्पिल के रूप में सीढ़ी के चरण बनाती हैं। यह भी स्थापित किया गया है कि A हमेशा दो हाइड्रोजन बांडों का उपयोग करके T से जुड़ा होता है, और G तीन समान बांडों द्वारा C से जुड़ा होता है। इस घटना को "चयनात्मकता और संपूरकता का सिद्धांत" नाम दिया गया था।
संरचनात्मक संगठन के स्तर
सर्पिल की तरह मुड़ी हुई एक पॉलीन्यूक्लियोटाइड श्रृंखला एक प्राथमिक संरचना होती है जिसमें 3',5'-फॉस्फोडाइस्टर बंधन से जुड़े मोनोन्यूक्लियोटाइड्स का एक निश्चित गुणात्मक और मात्रात्मक सेट होता है। इस प्रकार, प्रत्येक श्रृंखला में 3' सिरा (डीऑक्सीराइबोज) और 5' सिरा (फॉस्फेट) होता है। जिन क्षेत्रों में आनुवंशिक जानकारी होती है उन्हें संरचनात्मक जीन कहा जाता है।
डबल हेलिक्स अणु द्वितीयक संरचना है। इसके अलावा, इसकी पॉलीन्यूक्लियोटाइड श्रृंखलाएं प्रतिसमानांतर हैं और श्रृंखलाओं के पूरक आधारों के बीच हाइड्रोजन बांड द्वारा जुड़ी हुई हैं। यह स्थापित किया गया है कि इस हेलिक्स के प्रत्येक मोड़ में 10 न्यूक्लियोटाइड अवशेष होते हैं, इसकी लंबाई 3.4 एनएम है। यह संरचना वैन डेर वाल्स इंटरेक्शन बलों द्वारा भी समर्थित है, जो प्रतिकारक और आकर्षक घटकों सहित एक ही श्रृंखला के आधारों के बीच देखी जाती हैं। इन बलों को पड़ोसी परमाणुओं में इलेक्ट्रॉनों की परस्पर क्रिया द्वारा समझाया जाता है। इलेक्ट्रोस्टैटिक इंटरैक्शन द्वितीयक संरचना को भी स्थिर करता है। यह धनात्मक रूप से आवेशित हिस्टोन अणुओं और ऋणात्मक रूप से आवेशित डीएनए स्ट्रैंड के बीच होता है।
तृतीयक संरचना हिस्टोन, या सुपरकोइलिंग के चारों ओर डीएनए स्ट्रैंड की घुमावदार है। पाँच प्रकार के हिस्टोन का वर्णन किया गया है: H1, H2A, H2B, H3, H4।
न्यूक्लियोसोम का क्रोमैटिन में मुड़ना एक चतुर्धातुक संरचना है, इसलिए कई सेंटीमीटर लंबा डीएनए अणु 5 एनएम तक मुड़ सकता है।

डीएनए के कार्य
DNA के मुख्य कार्य हैं:
- वंशानुगत जानकारी का भंडारण. प्रोटीन अणु में पाए जाने वाले अमीनो एसिड का क्रम उस क्रम से निर्धारित होता है जिसमें न्यूक्लियोटाइड अवशेष डीएनए अणु में स्थित होते हैं। यह जीव के गुणों और विशेषताओं के बारे में सारी जानकारी को एन्क्रिप्ट भी करता है।
- डीएनए वंशानुगत जानकारी को अगली पीढ़ी तक पहुंचाने में सक्षम है। यह प्रतिकृति-स्व-दोहराव की क्षमता के कारण संभव है। डीएनए दो पूरक श्रृंखलाओं में विभाजित होने में सक्षम है, और उनमें से प्रत्येक पर (पूरकता के सिद्धांत के अनुसार) मूल न्यूक्लियोटाइड अनुक्रम बहाल किया जाता है।
- डीएनए की मदद से प्रोटीन, एंजाइम और हार्मोन का जैवसंश्लेषण होता है।
निष्कर्ष
डीएनए की संरचना इसे आनुवंशिक जानकारी का संरक्षक बनने और इसे भविष्य की पीढ़ियों तक पहुंचाने की अनुमति देती है। इस अणु में क्या विशेषताएं हैं?
- स्थिरता. यह ग्लाइकोसिडिक, हाइड्रोजन और फॉस्फोडिएस्टर बांड के साथ-साथ प्रेरित और सहज क्षति की मरम्मत के तंत्र के कारण संभव है।
- प्रतिकृति की संभावना. यह तंत्र दैहिक कोशिकाओं में गुणसूत्रों की द्विगुणित संख्या को बनाए रखने की अनुमति देता है।
- आनुवंशिक कोड का अस्तित्व. अनुवाद और प्रतिलेखन की प्रक्रियाओं के माध्यम से, डीएनए में पाए जाने वाले आधारों का अनुक्रम पॉलीपेप्टाइड श्रृंखला में पाए जाने वाले अमीनो एसिड के अनुक्रम में परिवर्तित हो जाता है।
- आनुवंशिक पुनर्संयोजन की क्षमता. इस मामले में, जीन के नए संयोजन बनते हैं जो एक दूसरे से जुड़े होते हैं।
इस प्रकार, डीएनए की संरचना और कार्य इसे जीवित प्राणियों में एक अमूल्य भूमिका निभाने की अनुमति देते हैं। यह ज्ञात है कि प्रत्येक मानव कोशिका में पाए जाने वाले 46 डीएनए अणुओं की लंबाई लगभग 2 मीटर है, और न्यूक्लियोटाइड जोड़े की संख्या 3.2 बिलियन है।